Introducción
La Periodontitis es una enfermedad inflamatoria que afecta los tejidos de soporte del diente 1. Uno de los factores que desencadenan la enfermedad es la persistencia de la biopelícula formada en la superficie dental y en el margen de la encía 2.
En Colombia, de acuerdo con el Tercer Estudio Nacional de Salud Bucal, la prevalencia de pérdida de inserción periodontal fue del 50.2% y esto genera para el sistema de seguridad social cada vez más saturación debido a la alta morbilidad.
Porphyromonas gingivalis se asocia a la aparición y progresión de la periodontitis crónica y agresiva 3-5, aunque también puede ser aislada con frecuencia de pacientes sanos 6. Posee múltiples factores de virulencia 4,7,8, dentro de los cuales, las fimbrias mayores se consideran uno de los más importantes 7,9. Las fimbrias mayores están compuestas por subunidades de fimbrillina 10, se encuentran situados en la superficie celular, y le confiere a la bacteria la capacidad de adherirse a los tejidos de soporte del diente, de la película adquirida y a otras especies bacterianas durante la formación y consolidación de la biopelícula. El gen que codifica las fimbrias mayores se denomina FimA y se conocen 6 genotipos, (I, Ib, II, III, IV, V), en base a su secuencia de nucleótidos 10.
En sujetos con periodontitis los genotipos FimA II y FimA IV han sido identificados con mayor frecuencia en contraste con los sujetos sanos en los que el genotipo FimA I es el más frecuente 11-14.
Este estudio, descriptivo transversal buscó determinar la prevalencia de genotipos FimA de P. gingivalis aisladas de muestras subgingivales de sujetos sanos, con gingivitis y con periodontitis crónica y determinar su asociación con otros microorganismos periodontopáticos como Aggregatibacter actinomycetemcomitans, Treponema denticola y Tannerella forsythia.
Materiales y Métodos
Se tomaron muestras subgingivales de 151 sujetos, seleccionados por conveniencia, de acuerdo a los criterios de inclusión. La muestra estuvo conformada por 67 hombres y 84 mujeres con edades comprendidas entre 22-84 años, (43.4 ± 10.6 años), que asistieron a las clínicas de la Escuela de Odontología de la Universidad del Valle, de la Universidad Santiago de Cali y de una clínica dental privada en Cali - Colombia; estos sujetos tenían diferentes diagnósticos periodontales (25 sanos, 77 gingivitis y 49 con periodontitis). Los pacientes autorizaron su participación en el estudio por medio de la firma del consentimiento informado. Este estudio fue aprobado por el comité de ética en humanos de la Facultad de Salud de la Universidad del Valle, protocolo Nº 012-010. Los criterios de inclusión fueron: tener más de 15 dientes en la boca, que no hubiesen tomado antibióticos, corticoides y/o AINES por lo menos tres meses antes de ser incluidos en el estudio, sin enfermedades sistémicas y que no hubiesen recibido tratamiento periodontal y/o limpieza dental profesional al menos seis meses antes de participar en el estudio.
Examen periodontal y muestreo
Un examen periodontal completo fue realizado por dos periodoncistas previamente calibrados de acuerdo a los criterios diagnósticos de la AAP 1999. Las mediciones de la profundidad de bolsa y la pérdida de inserción clínica fueron registradas en seis puntos alrededor de cada diente. Las variables consideradas para el diagnóstico clínico fueron la profundidad de sondaje, la pérdida de inserción clínica, el porcentaje de sangrado y el número de dientes perdidos. Se consideraron como sujetos sanos aquellos que no tenían signos de inflamación, una profundidad al sondaje igual o inferior a 3 mm, sin sangrado al sondaje y sin pérdida de inserción clínica. Los pacientes con gingivitis presentaron signos clínicos de inflamación gingival y edema, sangrado al sondaje, sin pérdida de inserción clínica. Los pacientes con periodontitis presentaron inflamación, edema, sangrado al sondaje y una profundidad al sondaje de 4 mm o mas en al menos un diente (localizado) o más del 50% de los dientes (generalizada). Después de la eliminación de la placa supragingival con gasas estériles, dos puntas de papel estéril se introdujeron en las bolsas más profundas (>4 mm) en pacientes con periodontitis y en la superficie vestibular mesial de los primeros cuatro molares en sujetos sanos y pacientes con gingivitis, dejándolos en el surco durante un minuto. A partir de entonces, las puntas de papel se depositaron en tubos Eppendorf y fueron criopreservados a -20° C hasta su procesamiento 14-17.
Extracción de ADN y PCR
La extracción de ADN bacteriano de las puntas de papel se llevó a cabo a través de la adsorción a partículas de sílice, de acuerdo con el protocolo del Boom 1989, y el procedimiento de genotipificación de P. gingivalis se realizó mediante PCR, utilizando cebadores publicados previamente 16-18. En primer lugar, la extracción de ADN se confirmó mediante el uso de cebadores genéricos específicos para el gen 16S rRNA bacteriano; a continuación, la presencia de ADN de P. gingivalis se confirmó con cebadores específicos 16-18 y, a partir de entonces se realizó una PCR con los cebadores para cada genotipo de FimA: I, Ib, II, III, IV, V, que han sido ampliamente reportados en la literatura 11,14-17.
El ADN de las cepas de P. gingivalis ATCC 33227 (FimA I), W83 (FimA IV), ATCC33279 (FimA Ib) fue utilizado como control positivo de estos genotipos 14. Para los controles positivos de los genotipos FimA II y FimA III, se utilizaron los aislados clínicos número 486 y 723, respectivamente, que fueron tipificadas y donados por el laboratorio de microbiología en la Universidad del Bosque 13.
Para estudiar los genotipos de FimA la PCR se realizó en un termociclador (marca Axygen) con el siguiente programa de amplificación: desnaturalización inicial a 95° C durante 5 min, seguido de 36 ciclos a 94° C durante 30 s, 60° C durante 30 s y 72° C durante 30 s, y una extensión final a 72° C durante 7 min. Además de los genotipos FimA de P. gingivalis, también se utilizaron las mismas muestras para identificar la presencia de otras bacterias periodontopáticas como: A. actinomycetemcomitans, T. denticola y T. forsythia a través de una sola ronda de PCR usando cebadores específicos para el gen 16S rRNA de cada uno de los tres microorganismos antes mencionados. Los productos de PCR se separaron por electroforesis en gel de agarosa y las bandas de ADN se tiñeron con SYBR SAFE y se visualizaron mediante luz UV en un trans-iluminador (marca Invitrogen). Los resultados fueron documentados a través de un registro fotográfico. La Identificación de los genotipos de FimA realizó de acuerdo con el tamaño molecular de las bandas de amplificación obtenidas, en comparación con el respectivo control positivo 14.
Estandarización y sensibilidad analítica de la PCR para la detección del gen FimA de P. gingivalis
Para determinar el límite de detección del gen FimA de P. gingivalis por la técnica de PCR, se llevaron a cabo diluciones seriadas de los cultivos de las cepas de los genotipos de P. gingivalis FimA II y FimA III (aislados clínicos previamente tipificados), FimA Ib (cepa ATCC33279). FimA IV (cepa W83), FimA I (cepa ATCC 33227), cepas que definimos como control para el propósito de este estudio. Además, para determinar la exactitud del procedimiento de PCR para detectar cada uno de los genotipos de FimA a partir de muestras clínicas de fluido gingival crevicular (GCF), los controles también fueron inoculadas experimentalmente con muestras de GCF negativos para P. gingivalis con cada una de las diluciones de las cepas de control de FimA y se realizó el procedimiento de PCR.
Especificidad de la PCR para la detección del gen FimA de P. gingivalis
Para determinar la especificidad de los cebadores utilizados en la PCR para detectar los diferentes genotipos de FimA de P. gingivalis, se realizó la prueba de PCR, optimizado para cada uno de los genotipos de FimA (I, II, II, IV, y Ib). La reactividad cruzada se estableció frente a otras bacterias periodontopáticas como A. actinomycetemcomitans (D11-s1), T. denticola (ATCC 43056), y T. forsythia (ATCC 43037).
Análisis estadístico
Se utilizó la prueba estadística del Chi cuadrado de Pearson para comparar las frecuencias de los genotipos FimA; para analizar las frecuencias de los otros periodontopatógenos, se aplicó la prueba exacta de Fisher, dado que el tamaño de la muestra fue inferior a 20. Las variables cuantitativas, como CAL (pérdida de inserción clínica), fueron analizadas con la prueba T de Student y las variables cuantitativas como PD (sondaje profundidad), BOP (sangrado al sondaje), número de dientes y edad se analizaron mediante el test de Wilcoxon y la prueba de Mann-Whitney. La selección de las pruebas utilizadas en las variables cuantitativas se basó en pruebas anteriores de distribución Normal.
Resultados
Sensibilidad y Especificidad de la PCR para el gen FimA de P. gingivalis
El límite de detección del gen FimA se determinó hasta 50 células bacterianas en las diluciones realizadas a partir de un cultivo puro de cada cepa de P. gingivalis disponible. Sin embargo, el límite de detección aumentó hasta 500 células cuando se inocularon las diluciones bacterianas en GCF. Este último se puede atribuir a la presencia de DNAasas presentes en el GCF. Se encontró una alta especificidad entre los cebadores utilizados y las cepas representativas de cada uno de los genotipos de FimA estudiados.
Frecuencia de P. gingivalis
Un total de 85 sujetos fueron positivos para P. gingivalis (56.3%), (57.6% en mujeres y 42.3% en hombres). La frecuencia de P. gingivalis por diagnóstico fue del 52.0% en sujetos sanos, 59.7% para pacientes con gingivitis y 53.1% para pacientes con periodontitis (Tabla 1).
| Genotyipos | Diagnostico periodontal | ||||||
|---|---|---|---|---|---|---|---|
| Sano | Gingivitis | Periodontitis | p value* | ||||
| **n | (%) | n | (%) | n | (%) | >0.050 | |
| P. gingivalis | 13 | 52.0 | 46 | 59.7 | 26 | 53.1 | 0.681 |
| FimA I | 2 | 15.4 | 12 | 26.1 | 3 | 11.5 | 0.301 |
| FimA II | 6 | 46.2 | 25 | 54.3 | 15 | 57.7 | 0.792 |
| FimA III | 2 | 15.4 | 2 | 4.3 | 2 | 7.7 | 0.386 |
| FimA IV | 1 | 7.7 | 6 | 13.0 | 0 | 0.0 | 0.154 |
| FimA Ib | 3 | 12.0 | 15 | 19.5 | 2 | 4.1 | 0.057 |
| FimA V | 0 | 0.0 | 0 | 0.0 | 0 | 0.0 | |
| No tipificable | 5 | 38.0 | 10 | 21.7 | 8 | 30.7 | 0.428 |
| fimA I, II, Ib | 1 | 7.7 | 8 | 17.4 | 0 | 0.0 | 0.066 |
| fimA III, Iv, Ib | 1 | 7.7 | 0 | 0.0 | 0 | 0.0 | 0.079 |
| fim A I, III, Ib | 1 | 7.7 | 0 | 0.0 | 0 | 0.0 | 0.079 |
| fimA II, IV | 0 | 0.0 | 2 | 4.3 | 0 | 0.0 | 0.377 |
| fimA I, II | 0 | 0.0 | 1 | 2.1 | 2 | 4.3 | 0.406 |
| fim A III, Ib | 0 | 0.0 | 0 | 0.0 | 1 | 2.1 | 0.350 |
| fim A III, IV | 0 | 0.0 | 1 | 2.1 | 0 | 0.0 | 0.616 |
| fim A I, Ib | 0 | 0.0 | 4 | 8.7 | 1 | 3.8 | 0.375 |
| *Chi cuadrado de Pearson **Los valores de n no suman el total que se muestra ya que el mismo sujeto puede expresar diversos tiempo genotipos | |||||||
Frecuencia y distribución de los genotipos de FimA de P. gingivalis
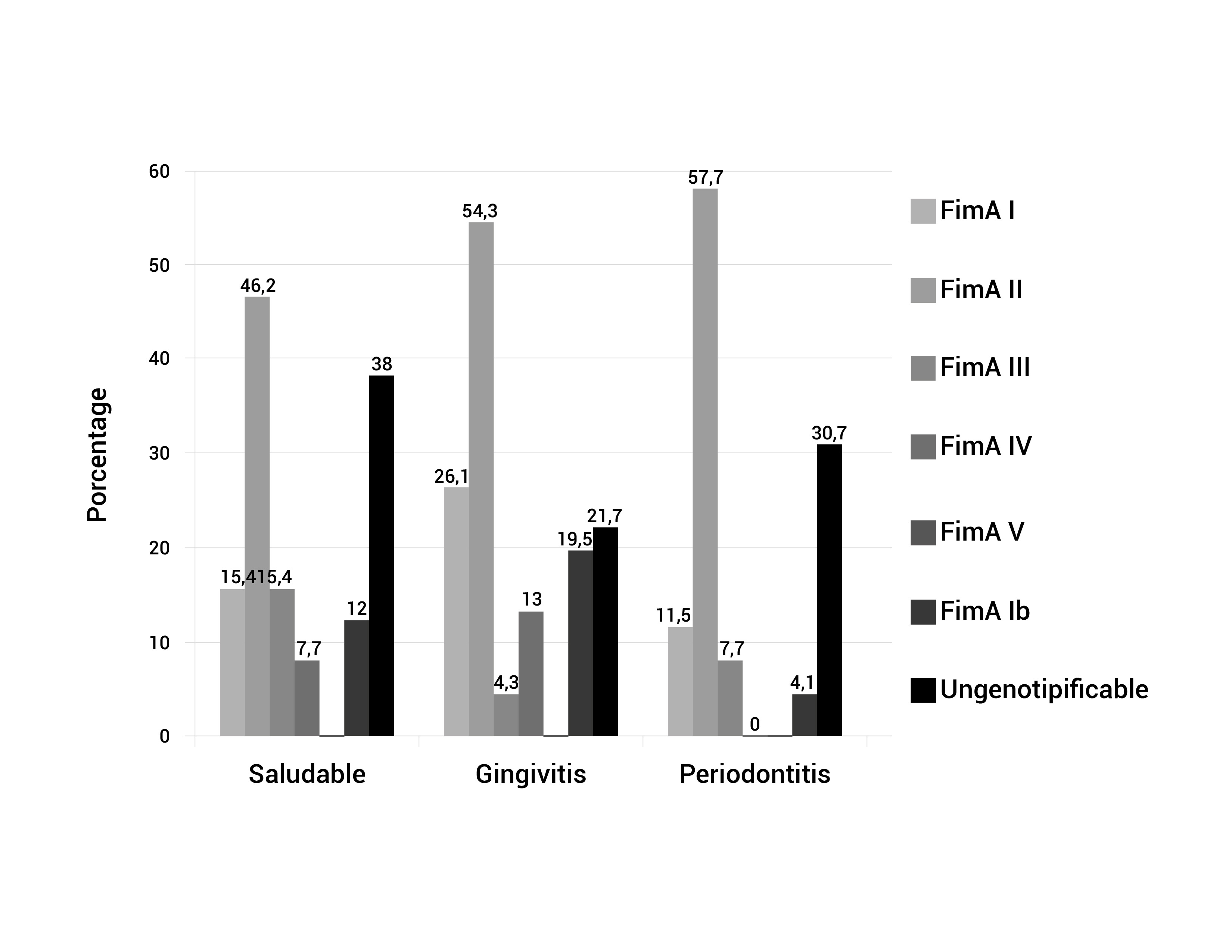
El genotipo mas frecuente fue FimA II (54.1%), seguido por FimA Ib (23.5%), FimA I (20.0%), FimA IV (8.2%), y FimA III (7.1%). No se encontraron muestras positivas para el genotipo FimA V. De los 85 pacientes positivos para P. gingivalis, 23 (27%) fueron negativos para los 6 genotipos estudiados, estas muestras se designaron como genotipos desconocidos o no genotipificables (Tabla 1). En los tres grupos los pacientes sanos, con gingivitis y periodontitis, los porcentajes de los genotipos fimA II fueron 46.2%, 54.3%, y 57.7%, respectivamente (Tabla 1, Fig. 1).
El genotipo FimA II presentó una tendencia a ser mas frecuente en los diagnósticos periodontales mas severos, estando alto en pacientes con periodontitis moderada y severa (AAP 1999), aunque no hubo diferencias estadísticamente significativas (p= 0.813). También se analizó la prevalencia y distribución de los diferentes genotipos de FimA de acuerdo a las variables clínicas, encontrándose que los genotipos FimA II y FimA III estuvieron relacionados con mayor PD por sitio, el genotipo FimA II estuvo relacionado con mayor CAL y mayor pérdida de dientes, mientras el genotipo FimA III estuvo relacionado con mayor BOP (Tabla 2). Once muestras fueron positivas para 3 genotipos y 12 muestras para 2 genotipos diferentes (Tabla 1).
| Genotipos | Variables clinicas* | |||
|---|---|---|---|---|
| PD | CAL | BOP | NoDi | |
| FimA I | 4.3 | 4.2 | 55.8 | 25.1 |
| FimA II | 4.6 | 4.5 | 53.4 | 23.2 |
| FimA III | 4.6 | 4.0 | 63.5 | 24.0 |
| FimA IV | 4.4 | 4.3 | 61.8 | 23.4 |
| FimA Ib | 4.2 | 4.0 | 56.0 | 24.0 |
| *Los datos son la media PD: sondeo sitio profundidad de la bolsa (mm) CAL: CAL: sitio de la pérdida de inserción clínica (mm) BOP: sangrado al sondaje (%) NoDi: número de dientes | ||||
Prevalencia de otros periodontopatogenos
La prevalencia de T. forsythia fue del 65.3% en pacientes con periodontitis, 68.8% en pacientes con gingivitis y 56.0% en pacientes sanos. La prevalencia de T. denticola en pacientes sanos, con gingivitis y periodontitis fue del 40.0%, 58.4% y 40.8% respectivamente y para A. actinomycetencomitans fue del 8.0% en sanos, 20.8% en pacientes con gingivitis y 14.2% en pacientes con periodontitis (Tabla 3).
| Especies* | ||||
|---|---|---|---|---|
| A. actinomyc** | T. denticola | T. forsythia | ||
| Diagnostico periodontal | Sano | 8.0 | 40.0 | 56.0 |
| Gingivitis | 20.8 | 58.4 | 68.8 | |
| Periodontitis | 14.2 | 40.8 | 65.3 | |
| P .gingivalis | 64.0 | 74.7 | 72.7 | |
| Genotipos FimA | ||||
| FimA I | 31.2 | 21.4 | 22.2 | |
| FimA II | 68.8 | 55.3 | 52.8 | |
| FimA III | 6.7 | 7.1 | 5.5 | |
| FimA IV | 6.7 | 8.9 | 6.94 | |
| *Porcentaje **A. actinomycetemcomitans |
||||
Co-infección de P. gingivalis y genotipos de FimA con A. actinomycetemcomitans, T. denticola, y T. forsythia
Se observó una alta correlación entre P. gingivalis, T. denticola, y T. forsythia, (p= 0.000). La co-infección de estos microorganismos fue observada en altos porcentajes para el genotipo FimA II con frecuencias en las muestras positivas para A. actinomycetemcomitans de 68.7%, T. denticola 55.3%, y T. forsythia 52.8%, aunque esta relación no fue estadísticamente significante. (Tabla 3).
Discusión
Frecuencia de P. gingivalis
Los resultados de este estudio en la frecuencia de P. gingivalis difieren de los reportados por Amano et al. 14,15, Guo et al. 19, Wu et al. 22, Zhao et al. 23, pero son similares a los reportados por Beikler 20 y por Missailidis et al 16.
La alta prevalencia de P. gingivalis es común en la población colombiana, de acuerdo a lo reportado por Botero et al. 24, Lafaurie et al. 25, y Mayorga et al 26. Estos hallazgos pueden ser debidos a las condiciones de sanidad, pobreza y vivienda. Los pacientes incluidos en este estudio viven con un aproximado de $350 US dólares por mes. Esto dificulta el acceso a los servicios dentales, el empeoramiento de la periodontitis por el retraso o la falta de tratamiento dental, favoreciendo quizás, la transmisión horizontal y vertical de patógenos periodontales.
Frecuencia y distribución de los genotipos de FimA de P. gingivalis
En general, el genotipo mas frecuente fue FimA II, el cual se detectó en 46 muestras (54.1%).
En los pacientes sanos los genotipos mas frecuentes fueron FimA II seguido por FimA I, estos resultados son diferentes a los publicados en estudios previos que reportaron bajas frecuencias de P. gingivalis en pacientes sanos 16,20,21, así como bajas frecuencias del genotipo FimA II y altas frecuencias de los genotipos FimA I, FimA III, y FimA V en este tipo de pacientes 14,16,22,23, considerando estos últimos genotipos como poco virulentos (Tabla 1, Fig. 1).
Vale la pena considerar la importancia de realizar estudios en los que se determine la expresión de los genotipos, por ejemplo a través de PCR en tiempo real, los cuales permitirán establecer la relación entre el fenotipo y el genotipo. Otro asunto importante para tener en cuenta es la posibilidad de que los genotipos mas virulentos se encuentran en los estadios iniciales de la enfermedad periodontal donde inician los procesos de infección y colonización así como el estímulo de la respuesta inmune e inflamatoria, la cual después del tiempo, puede generar daño y destrucción del tejido, y los resultados hallados con relación a los genotipos encontrados mas frecuentemente en el grupo de sanos y de pacientes con gingivitis, puede estar asociado a la carga bacteriana en estos estadios iniciales; sin embargo se requieren mas estudios para probar esta hipótesis, estudios que incluyan PCR en tiempo real para cuantificar y establecer diferencias entre los diferentes diagnósticos periodontales.
Los resultados de los grupos de pacientes con gingivitis y periodontitis, referentes a la alta prevalencia del genotipo FimA II, específicamente en gingivitis generalizada, periodontitis moderada y periodontitis severa, coinciden con los estudios de Japón 15,16,18, China 19,23, Alemania 20, Noruega, Estados Unidos, Canadá, Holanda 21, Brasil 11,16, México 17, Colombia 13. El Segundo genotipo mas común en estos dos grupos (gingivitis y periodontitis) fue FimA I, el cual ha sido asociado en diferentes estudios a individuos sanos, en el presente estudio se encontró una frecuencia de 26.1% y 11.5%, respectivamente (Tabla 1), lo cual es muy similar a lo reportado por Missailidis et al. 16, y Davila et al 17.
En nuestro estudio no hubo diferencias estadísticamente significativas en la distribución de los genotipos por diagnóstico periodontal. Moon et al., publicaron la detección de una reacción cruzada entre los cebadores específicos para la detección del genotipo FimA II, con los genotipos FimA Ib, cabe aclarar que los estudios previos, reportados en este trabajo, utilizaron estos cebador. Este hallazgo conlleva al supuesto que dicha reacción cruzada, podría conllevar a errores en los resultados previamente reportados 27. El mismo grupo publicó otro estudio en 2013 donde probaron los nuevos primers para FimA II en población Coreana, encontrando una distribución de 44.5% para pacientes sanos y 67.3% para pacientes con periodontitis, (sin diferencias estadísticamente significativas), concluyendo que los resultados de previos estudios deben ser revisados pues conllevaron a una sobre estimación del riesgo relativo para el genotipo FimA II, sin embargo, dada la patogenicidad de estos genotipos, demostrada en experimentos in vitro, los autores sugieren que los sujetos sanos portadores del genotipo FimA II, tienen un riesgo incrementado de desarrollar enfermedad periodontal 28. Los resultados del estudio en Corea son muy similares a nuestros hallazgos.
Todas las muestras positivas para P. gingivalis fueron negativas para en genotipo FimA V (Tabla 1). Estos resultados coinciden con los de Mssailidis et al. 16, y por Enersen et al. 29, pero difieren de los resultados de Japón 18, y de China 23 donde reportaron prevalencias de 16 a 29%; sin embargo se ha reportado muy baja prevalencia de este genotipo (1 to 3.9%) en estudios de Alemania 20 y Noruega 21.
Cabe resaltar que en el desarrollo de este estudio, la cepa control del genotipo FimA V de P. gingivalis no estuvo disponible y aunque se realizó la PCR usando primers específicos para este genotipo, ninguna muestra resultó positiva. La falta de la cepa control para el genotipo FimA V fue una limitación del estudio.
Acerca de la relación con las variables clínicas, se encontró que el genotipo FimA III fue el mas frecuente en los pacientes con valores altos en el promedio de PD, CAL y BOP. No obstante, cuando se compara la relación de los genotipos con la profundidad al sondaje y la pérdida de inserción en los sitios de toma de las muestras (Tabla 2), el genotipo FimA II fue el mas relacionados con mayor PD y mayor CAL. Una tendencia similar fue encontrada en estudios previos 11,14,29.
La presencia de más de un genotipo ha sido ampliamente reportada en la literatura 16,30. En el presente estudio, 23 muestras, en su mayoría distribuidas en el grupo de gingivitis, presentaron frecuencias del 9.4% del total para dos genotipos y del 9.4% para tres genotipos (Tabla 1). Esta distribución heterogénea de las poblaciones, puede estar asociado a diferencias étnicas discutidas anteriormente 16,17,20.
Prevalencia de microorganismos periodontopatogenos: A. actinomycetemcomitans, T. denticola, y T. forsythia
Este estudio encontró una alta prevalencia de tres microorganismos como A. actinomycetemcomitans, T. denticola, y T. forsythia, respecto al total de la muestra, siendo mas frecuentes en los grupos de gingivitis y periodontitis comparado con los individuos sanos, lo cual coincide con otros estudios en Colombia como los de Botero et al. 24 y Lafaurie et al 25.
Co-infección de los genotipos de Fim A de P. gingivalis con T. denticola, T. forsythia, y A. actinomycetemcomitans
En este estudio, se encontró una asociación positiva entre los tres microorganismos del complejo rojo, tal como lo descrito por Socransky et al., 31 y corroborado por otros investigadores 23-25 (Tabla 3). El estudio también encontró un alto porcentaje de muestras positivas para P. gingivalis y A. actinomycetemcomitans; estos dos microorganismos han sido considerados como importantes factores etiológicos durante el inicio y progresión de la enfermedad periodontal 32. De las muestras positivas para P. gingivalis y los otros tres microorganismos estudiados, la mayoría corresponden al genotipo FimA II, lo cual podría indicar una alta relación de coinfección de este genotipo con T. denticola, T. forsythia y A. actinomycetemcomitans. Estos resultados son similares a los obtenidos por Zhao et al 23.
Conclusions
El genotipo FimA II de P. gingivalis fue el mas frecuente de los aislados de bolsas periodontales y esto estuvo asociado a la severidad de la enfermedad periodontal.
Porphyromonas gingivalis se asoció con T. denticola y T. forsythia, y el genotipo FimA II fue mas frecuente en pacientes que presentaron co-infección con estos periodontopatógenos.