Introducción
El gen NAT2 codifica para la enzima Arilamina N-acetiltransferasa 2 (NAT2), la cual interviene en la fase II de la detoxificación metabolizando xenobióticos y componentes que contienen aminas aromáticas por N- u O-acetilación; además, metaboliza antibióticos como la Isoniacida que se utiliza en el tratamiento contra la Tuberculosis 1-3.
El gen NAT2 se encuentra localizado en el brazo corto del cromosoma 8 (8p22) y se han descrito hasta la fecha 59 variantes alélicas en diferentes poblaciones humanas, las cuales presentan entre una y cuatro sustituciones de nucleótidos en las posiciones 191, 282, 341, 434, 481, 590, 803, 845, y 857; siete de estas originan un cambio en el aminoácido codificado con excepción de las posiciones 282 y 481 4-7.
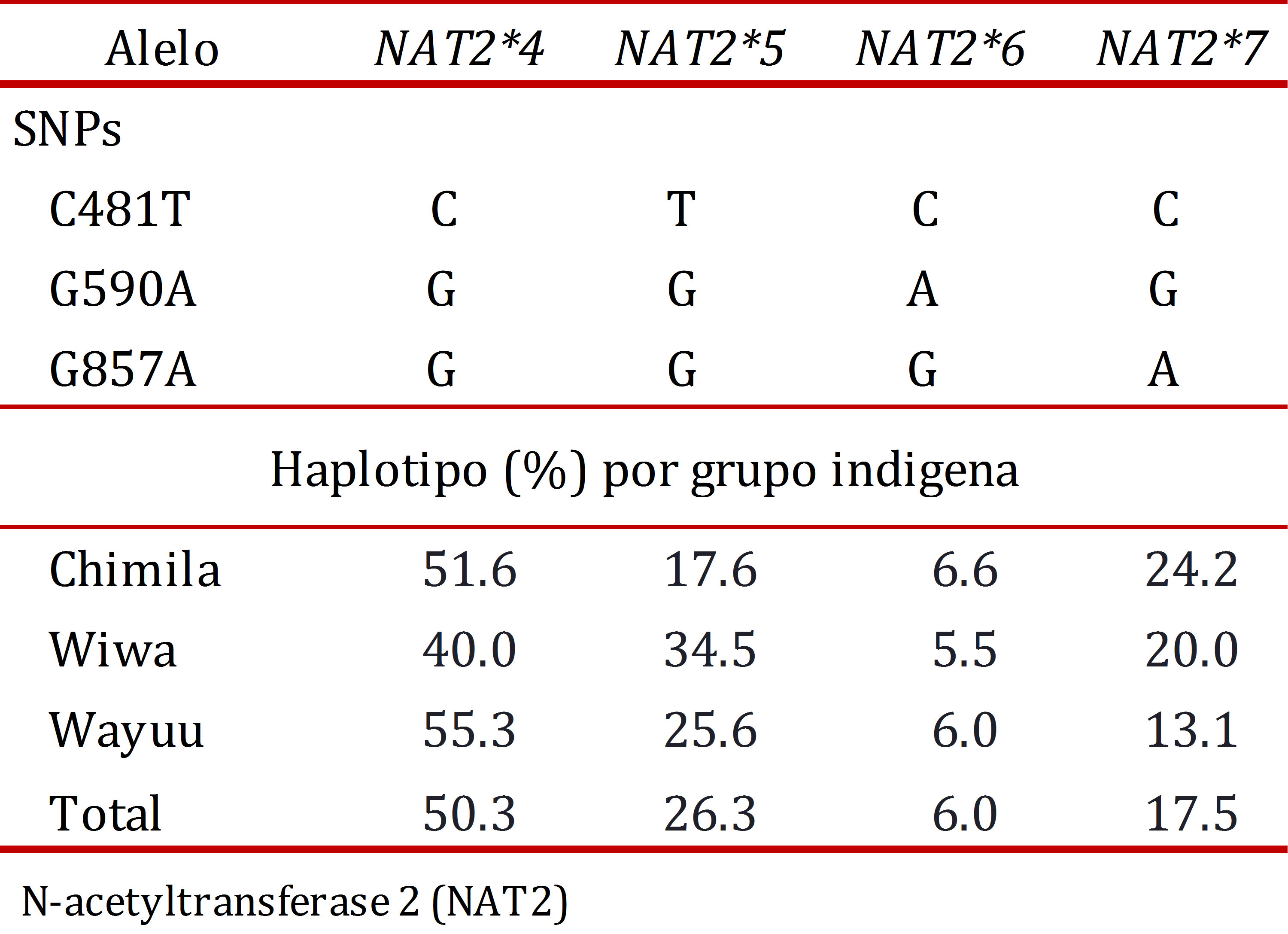
El gen NAT2, tiene cuatro alelos principales: NAT2*4, es el alelo normal o wild-type mientras que NAT2*5, NAT2*6 y NAT2*7 son los alelos mutados determinados por la presencia de los polimorfismos tipo SNP en las posiciones 481C>T, 590G>A y 857G>A respectivamente. El cambio nucleotídico en la posición 481 no origina cambio en el aminoácido codificado, en la posición 590 se origina un cambio del aminoácido Arginina (Arg) por Glutamina (Gln) y en la posición 857 un cambio del aminoácido Glicina (Gly) por Acido glutámico (Glu) 8.
Dependiendo de la actividad de la enzima, se han determinado 3 tipos de acetiladores: los acetiladores rápidos tienen dos alelos normales y su genotipo es NAT2*4/*4; los acetiladores intermedios tienen un alelo normal y uno mutado con posibles genotipos NAT2*4/*5, NAT2*4/*6 y NAT2*4/*7; y los acetiladores lentos tienen dos alelos mutados y posibles genotipos NAT2*5/*5, NAT2*5/*6, NAT2*5/*7, NAT2*6/*6, NAT2*6/*7 y NAT2*7/*7 (Tabla 1) 4,9.
La frecuencia del fenotipo acetilador varía de acuerdo a los grupos étnicos; el fenotipo acetilador lento se presenta en caucásicos en un 40-60%, en Afroamericanos 60%, en orientales 10-20%, en esquimales 5% y en poblaciones mediterráneas en un 90% 7,10.
Estudios realizados en poblaciones amerindias de Argentina y Paraguay, han reportado una frecuencia de más del 40% para el alelo NAT2*4 (42.9-80.0%); así mismo, el alelo NAT2*5 se encontró con frecuencias elevadas entre 31.2% y 50.0% en los grupos Mapuche y Tehuelche (Patagonia Argentina) y los grupos Lengua y Ayoreo (Paraguay). El alelo NAT2*6 estuvo presente en un 3% en Wichi y en más del 12% en las poblaciones Jujuy, Mapuche y Tehuelche mientras que el alelo NAT2*7 mostró una alta variabilidad del 4.5% al 42.9% en las diferentes poblaciones 10. Por su parte, Jorge-Nebert et al., reportaron en las poblaciones de Ngawbe y Embera, una frecuencia de 2.4% y 9.9% para el alelo NAT2*5, de 0% y 3.7% para el alelo NAT2*6 y de 23.3% y 22.8% para el alelo NAT2*7, respectivamente 11. En un estudio reciente en las poblaciones nativas de Brasil, encontraron en el grupo amerindio Tupinamba, una frecuencia de 43.3% para el alelo NAT2*5 y NAT2*6 y una frecuencia del 10% para el alelo NAT2*7 12.
Debido a que los polimorfismos tipo SNPs en las posiciones 481, 590 y 857 son la causa del 95% de alelos con baja actividad enzimática 1,4,9 y su determinación permite definir el estado acetilador de los individuos, el objetivo de la presente investigación consiste en estudiar estos polimorfismos en los grupos indígenas Chimila, Wiwa y Wayúu representativos del Caribe Colombiano para determinar las frecuencias alelicas y genotípicas, y así determinar qué tipo de acetiladores se encuentran presentes en estas poblaciones.
Materiales y Métodos
Población
En el presente estudio se incluyeron 202 individuos pertenecientes a tres grupos indígenas que habitan la Región Caribe de Colombia. Para la toma de las muestras se tuvo en cuenta los rasgos fenotípicos que caracterizan a las poblaciones indígenas (delgados, cabello liso negro, ojos color negros, color de piel rojo y estatura mediana), su ubicación geográfica y que mantuvieran sus tradiciones y ritos de acuerdo a sus conductas socio-culturales de tiempos prehispánicos y baja grado de consanquinidad. El grupo indígena Ette ennaka o Chimila (gente propia), conocidos en la literatura etnográfica como los Simiza (Preuss, 1926), Chimíle o Simza (Ortiz, 1965), o Shimizya (Loukotka, 1968)(referenciados en 13), se ubican en la región natural de las llanuras centrales del Departamento del Magdalena, en el asentamiento Naara Kajmanta en la vereda de Puerto Mosquito estribaciones de la Sierra Nevada de Santa Marta, en el municipio de Sabanas de San Ángel, en el resguardo llamado Issa Oristunna (Tierra de la Nueva Esperanza). Hablan el idioma ette taara (lengua de la gente), el cual corresponde a la familia lingüística Chibcha. Su población se estima en 910 individuos 13-16.
El grupo indígena Wiwa, también conocidos como arsario, guamaca, malayo, sanjá, dumana, actualmente pertenecen a la población el Encanto (Gotsezhi) del corregimiento de Guachaca en las estribaciones de la Sierra Nevada de Santa Marta, Departamento del Magdalena. Su lengua materna es damana de la familia lingüística Chibcha y su población alcanza 13,627 individuos 15-17. El grupo indígena Wayúu (señor, hombre poderoso), también conocidos como wayu, uáira, waiu, se caracterizan por tener nariz ancha, ojos color negro, cabello negro y lacio, estatura mediana; son robustos y corpulentos. Habitan en la parte media y alta de la península de la Guajira sobre el mar Caribe, en el municipio de Manaure, Departamento de la Guajira y su lengua se denomina wayuunaiki perteneciente a la familia lingüística Arawak. La población estimada para Colombia es de 149,827 individuos 15,18.
Esta investigación fue evaluada y aprobada por el Comité de Bioética en Investigación de la Universidad Cooperativa de Colombia, Sede Santa Marta (resolución 024 de 2010). Todos los individuos participantes dieron su consentimiento informado para la toma de las muestras.
Genotipificación
Se tomó una muestra de sangre periférica de cada individuo en tubo con anticoagulante (EDTA); el ADN genómico, fue aislado de linfocitos por medio del Kit UltraCleanTM Blood (Mo-Bio). Los tres SNPs C481T (rs1799929), G590A (rs1799930) y G857A (rs1799931) fueron determinados siguiendo la técnica de PCR en tiempo real por el método de discriminación alélica TaqMan empleando los siguientes ensayos: C_1204092_20, C_1204091_10, C_572770_20, respectivamente, en el equipo ABI PRISM 7500 Real-Time PCR Systems de Applied Biosystems, los cuales permiten detectar los cuatro alelos principales NAT2*4, NAT2*5, NAT2*6, NAT2*7 19.
Las condiciones de amplificación para un volumen final de reacción de 25 μL fueron las siguientes: 20 ng de ADN genómico, 12.5 μL de TaqMan Universal PCR Master Mix que contiene AmpliTaq Gold DNA polimerasa, AmpErase(r) Uracil N-glicosilasa (UNG), desoxinucleotidos (dNTPs) con dUTP, Referencia Pasiva (ROX) y Buffer; 1.25 μL de 20x Drug Metabolism Genotyping Assay Mix (especifico para cada polimorfismo) que contiene 18 μM de cada primer y 4 μM de cada sonda (VIC/FAM). Todos los ensayos fueron realizados siguiendo un mismo protocolo de amplificación y detección que inicia con un ciclo a 50º C por 2 min, un ciclo a 95º C por 5 min, 50 ciclos cada uno a 92º C por 10 s y un ciclo a 60º C por 90 s19. Los polimorfismos fueron determinados de acuerdo a las curvas de amplificación reconocidas a partir del marcaje para cada sonda (VIC/FAM).
Análisis estadístico
El análisis de frecuencias alélicas y genotípicas, se realizó usando la prueba de Chi cuadrado X2 con en el programa SPSS(r) (Statistical Package for the Social Science) para Windows(r) v.19.0. La comparación de la frecuencia de una sola mutación, de alelos y genotipos entre las poblaciones se realizó con el programa estadístico Arlequín(r) v.3.5. La frecuencia esperada de los genotipos fue calculada con la ecuación de Hardy-Weinberg (1= p2+2pq+q2). Finalmente, se realizó una comparación de las frecuencias alélicas y genotípicas encontradas en este estudio con las reportadas en la literatura.
Resultados
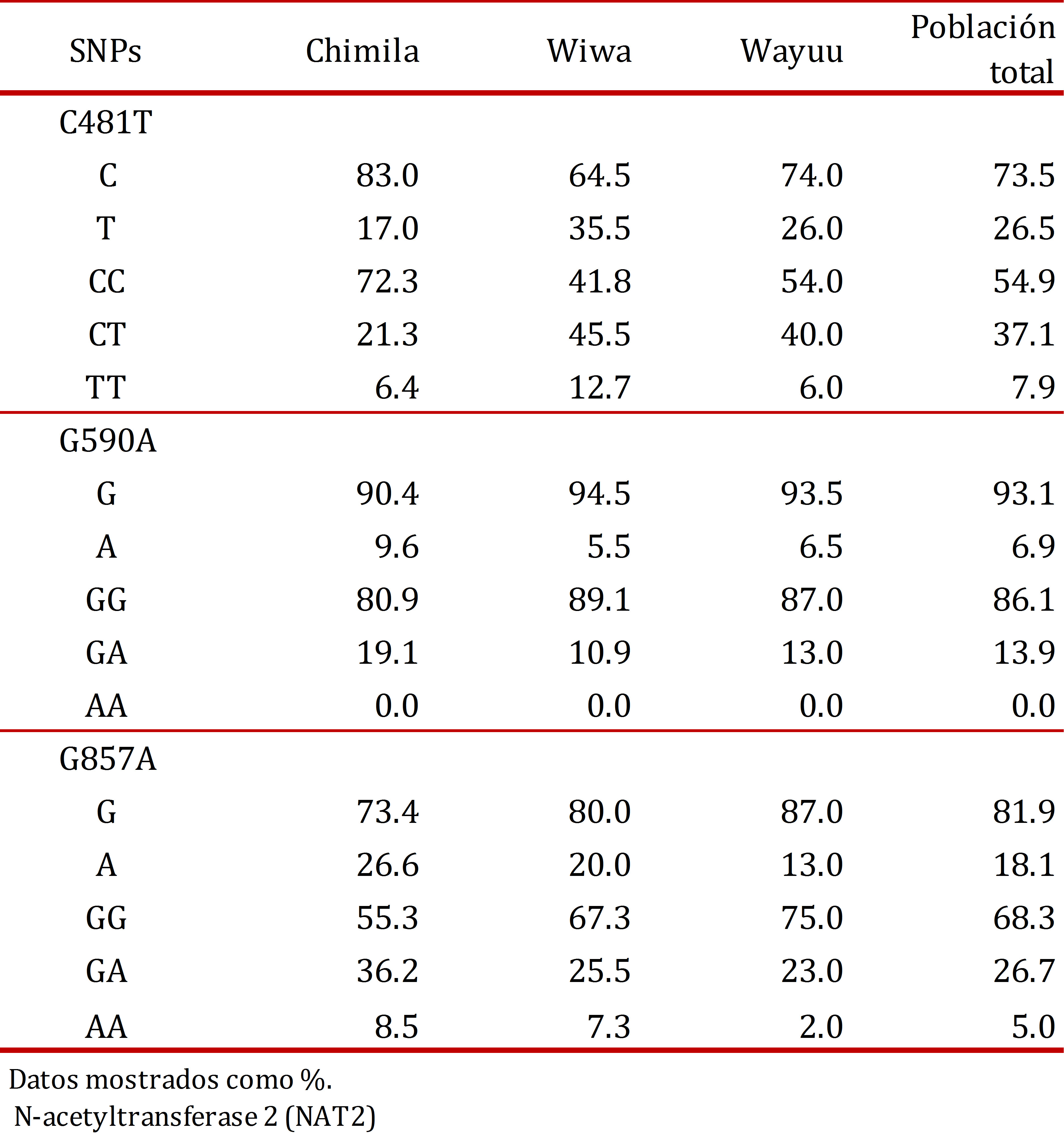
La población de estudio, estuvo conformada por 47 Indígenas Chimila (51% hombres y 49% mujeres), 55 Indígenas Wiwa (53% hombres y 47% mujeres) y 100 Indígenas Wayúu (11% hombres y 89% mujeres). Según los resultados obtenidos para cada SNP en la población total estudiada, el alelo 481T fue el más frecuente con 26.5%, seguido por el 857A con 18.1% y el menos frecuente fue 590A con 6.9%. Las frecuencias alélicas y genotípicas de los SNPs del gen NAT2 obtenidas para toda la población estudiada y por cada grupo indígena se encuentran descritas en la Tabla 2.
El alelo NAT2*4 (wild type) estuvo presente en el 50.3% de la población total, y más frecuente en el grupo Wayúu (55.3%). Entre los alelos mutados el NAT2*5 fue el más frecuente con 26.3% y el alelo NAT2*6 fue el menos frecuente con 6.0% en la población.
Con respecto a cada grupo estudiado, el SNP 481T fue el más frecuente en el grupo Wiwa con un 35.5%; el SNP 590A estuvo más frecuente en el grupo Chimila con 9.6%, y el SNP 857A mostró una alta frecuencia en el grupo Chimila y Wiwa con una frecuencia de 26.7% y 20.0% respectivamente. Para los alelos, el NAT2*5 mostró una alta frecuencia en el grupo Wiwa con 34.5% y el alelo NAT2*7 en el grupo Chimila con 24.2% (Tabla 3).
La distribución de los alelos del gen NAT2, estuvo en equilibrio de Hardy-Weinberg (p >0.05) en los tres grupos estudiados.
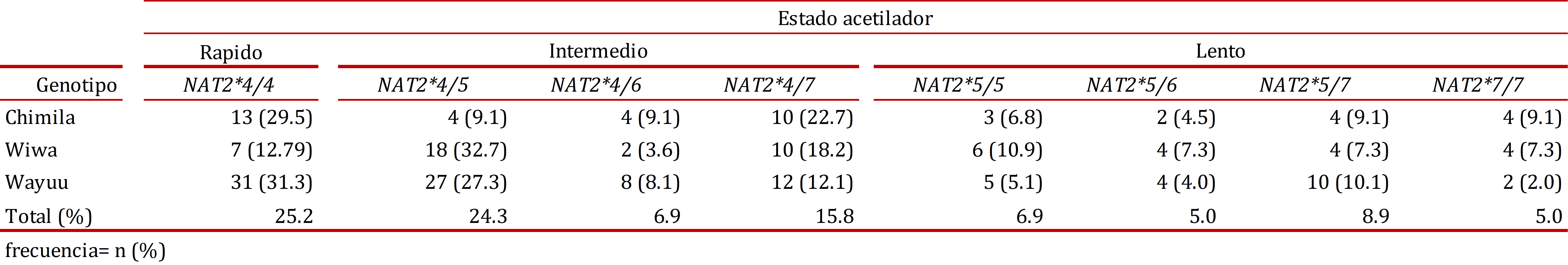
Según la combinación de alelos, ocho genotipos estuvieron presentes en la población analizada, los genotipos NAT2*6/*6 y NAT2*6/*7 no fueron encontrados. El genotipo NAT2*4/*4 asociado al estado acetilador rápido fue el más frecuente (25.2%) en los tres grupos estudiados con respecto a los otros genotipos.
En el grupo Wiwa el genotipo NAT2*4/5, acetilador intermedio, se presentó con una elevada frecuencia 32.7% con respecto al grupo Chimila (9.1%) y el grupo Wayúu (27.3%). Todos los genotipos que determinan el estado acetilador lento tuvieron una distribución similar (2.0%-11.0%) siendo más frecuente el genotipo NAT2*5/*5 con 10,9% en el grupo Wiwa, seguido del grupo Wayúu con 10.1%.
La frecuencia del estado acetilador rápido estuvo más alta en el grupo Wayúu (31.3%) y en el grupo Chimila (29.5%) cuando comparamos con el grupo Wiwa (12.7%). La distribución del estado acetilador intermedio es muy similar entre los tres grupos; para el estado acetilador lento observamos que en el grupo Wiwa la frecuencia es mayor (32.7%) con respecto a Chimila y Wayúu con 20.5% y 21.2% respectivamente (Tabla 4).
Discusión
El estudio consecutivo del polimorfismo del gen NAT2 en un gran número de poblaciones, ha mostrado la gran diversidad alélica y variación inter étnica de este gen; esto se refleja en los diferentes tipos de acetiladores presentes en las poblaciones analizadas.
La frecuencia del fenotipo acetilador varía de acuerdo a los grupos étnicos. El fenotipo acetilador lento se presenta en caucásicos en un 40-60%, hasta un 60% en Afroamericanos, 10-20% en Orientales, 5% en Esquimales y 90% en algunas poblaciones Mediterráneas. La frecuencia de acetiladores intermedios en Caucásicos es de 35.7%, en Japoneses es de 45.9% y en la población China es de 46.6% 20-22. Por el contrario, la frecuencia de acetiladores rápidos es de 25% en Caucásicos y 70% en Japoneses 23,24. Las poblaciones Caucásicas y Africanas tienen alta frecuencia de alelos NAT2*5 (28%) y bajas frecuencias de alelos NAT2*7 (7%), mientras que las poblaciones Asiáticas tienen baja incidencia de alelos NAT2*5 1,5,8,10.
Estudios realizados en poblaciones Amerindias de Argentina y Paraguay han reportado una frecuencia de más del 40% para el alelo NAT2*4 (42.9-80.0%); así mismo el alelo NAT2*5 se encontró con frecuencias elevadas entre 31.2% y 50.0% en los grupos Mapuche y Tehuelche (Patagonia Argentina) y los grupos Lengua y Ayoreo (Paraguay), respectivamente. Por su parte, el alelo NAT2*6 estuvo presente en 3% en Wichi y en más del 12% en las poblaciones de Jujuy, Mapuche y Tehuelche mientras que el alelo NAT2*7 mostró una alta variabilidad entre las diferentes poblaciones (4.5%- 42.9%) 10.
Por su parte, Jorge-Nebert et al., reportaron una frecuencia de 2.4% y 9.9% para alelo NAT2*5, mientras que NAT2*6 mostró una frecuencia de 0% y 3.7% y NAT2*7, 23.3% y 22.8% en las poblaciones de Ngawbe y Embera, respectivamente 11. En un estudio reciente en las poblaciones nativas de Brasil, se ha encontrado en el grupo amerindio Tupinamba, una frecuencia de 43.3% para el alelo NAT2*5 y NAT2*6; y una frecuencia del 10.0% para el alelo NAT2*7 12.
Los resultados obtenidos en los tres grupos analizados en este estudio, muestran como el patrón de las distribuciones alélicas y fenotípicas, presenta diferencias importantes a pesar de compartir una situación geográfica cercana y lenguajes similares (Chibcha, Wiwas y Chimilas). Esto, sin embargo, ha sido observado en el análisis de otros grupos indígenas de Sur y Centroamérica (Embera, Ngawbe, Tupinamba, entre otros) 11.
En los tres grupos estudiados, el alelo NAT2*4, presentó la mayor frecuencia alélica (Chimila: 51.6 %, Wiwa 40.0%, Wayúu: 55.3%), lo cual, no representa diferencia entre los mismos. El alelo NAT*6, mostró una distribución muy similar entre los tres grupos (Chimila: 6.6 %, Wiwa 5.5%, Wayúu: 6.0%). Diferencias inter-grupo se encontraron con relación al alelo NAT2*5 con un 17.5 % en Chimila comparado con 34.5 % en Wiwa y Wayúu 25.7%. En forma similar, el alelo NAT2*7, presentó una frecuencia menor en Wayúu (13.0%), comparado con 24.2 % en Chimila y 20.0% en Wiwa. La organización de los alelos en genotipos, nos permite inferir el estado acetilador de los diferentes grupos. En este sentido, de los tres grupos analizados el grupo Wiwa presentó un bajo porcentaje de acetiladores rápidos 12.7%, con respecto al grupo Chimila 29.5% y Wayúu con 31.3%, debido a que se presentó en la población en general una elevada frecuencia de individuos acetiladores lentos.
Cuando se comparan los resultados obtenidos con otros grupos indígenas (Tabla 5), el alelo NAT2*4 en las poblaciones estudiadas como en otras poblaciones de Panamá (Ngawbe y Embera)11 y Brasil (Amerindios), se presentó con mayor frecuencia con respecto a los alelos mutados NAT2*5, NAT2*6, NAT2*7. El alelo NAT2*5 se encontró en el grupo Wiwa con mayor frecuencia (34.5%) entre los grupos estudiados y mucho más alta cuando comparamos con los grupos Ngawbe y Embera, 2.4% y 9.2% respectivamente11; el alelo NAT2*6 se encontró con frecuencias más altas en todos los grupos estudiados al comparar con el grupo Embera (3.7%), mientras que en el grupo Ngawbe estuvo ausente. Por su parte, en el grupo Chimila (24.7%) y Wiwa (20.0%), el alelo NAT2*7 presentó frecuencias similares al comparar con las poblaciones nativas de Panamá (Ngawbe y Embera) 11,12.
Tabla 5.
Frecuencia de alelos del gen NAT2 en los grupos Indígenas Chimila, Wiwa, Wayúu y otras poblaciones.
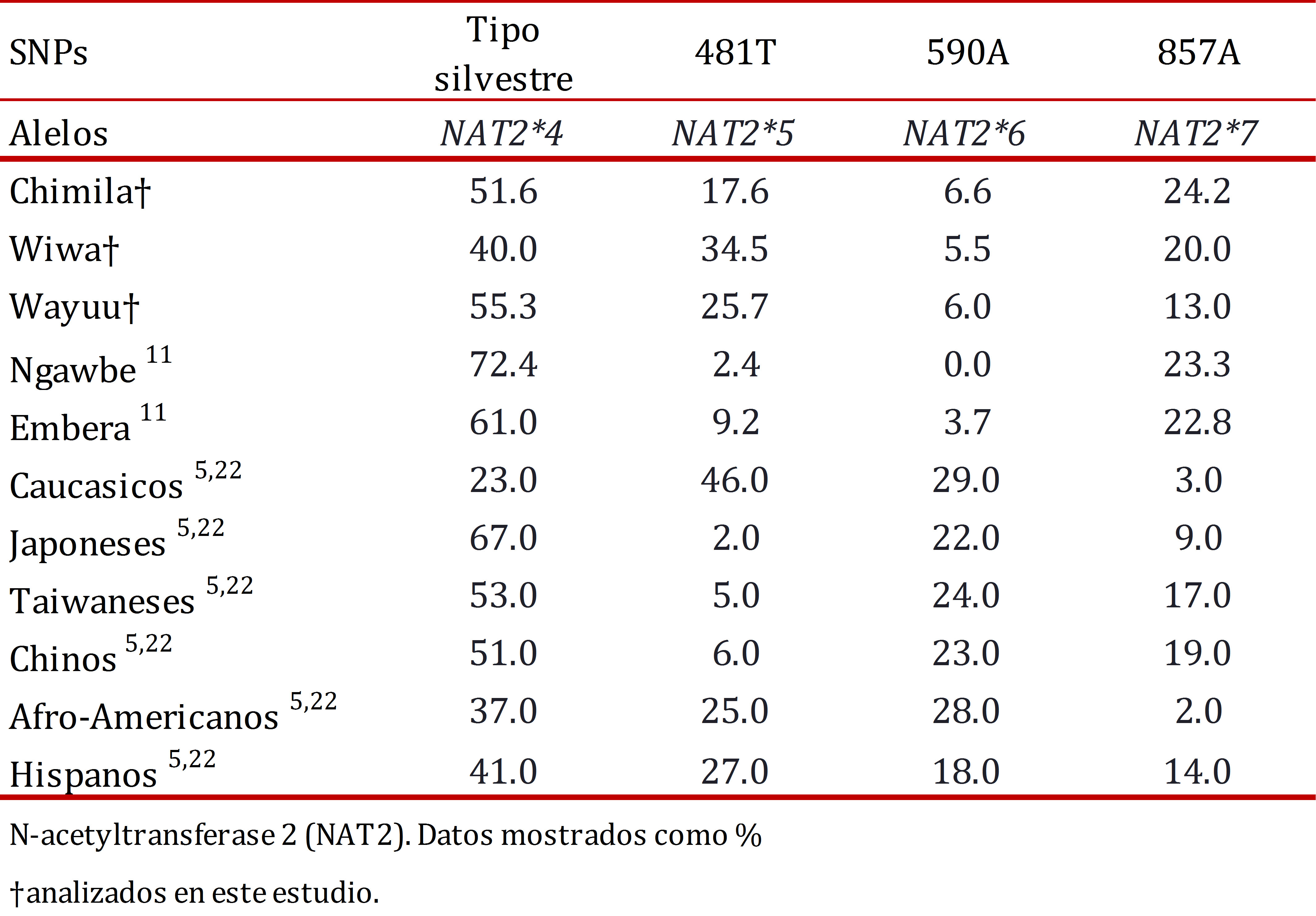
Con respecto al estado acetilador lento, se presentó con mayor frecuencia en los grupos estudiados aproximadamente en 50.0% cuando comparamos con las frecuencias reportadas para Ngawbe 7.6% y Embera 14.7% 11.
El estudio del polimorfismo del gen NAT2 en las poblaciones analizadas, muestra finalmente la gran variabilidad genética de estos grupos independiente de sus raíces lingüísticas y ubicación geográfica, debido posiblemente al bajo grado de mezclas entre las poblaciones, a lo cual, contribuye sus condiciones socioculturales y económicas. Estos estudios, son importantes por su aplicación al conocimiento de la Farmacogenómica de la población Caribe Colombiana y en especial por su utilidad en el tratamiento de la Tuberculosis con medicamentos que son metabolizados en forma específica por este gen y cuya prevalencia es alta en estas poblaciones.
Conclusión
La determinación de los Polimorfismos 481T, 590A, 857A presentes en el gen NAT2, permitió conocer la distribución alélica y diferencias farmacogenéticas de los tres grupos estudiados, Chimila, Wiwa y Wayúu; predecir el estado acetilador más frecuente que sería útil en la selección de medicamentos y dosis adecuada para el tratamiento de diversas enfermedades que son blanco de medicamentos metabolizados por la enzima N-acetil transferasa 2. Además, se podría deducir que la alta prevalencia de acetiladores lentos, podría tener un impacto en la alta incidencia de hepatotóxicidad inducida por medicamentos antituberculosos indicados en estas poblaciones.