Estudio preliminar de la susceptibilidad antimicrobiana y variabilidad genética de Mycobacterium tuberculosis en un área del Caribe colombiano
Jorge Miranda, Bacteriol.1, Rodrigo Ríos, Bacteriol.1, Andrea Clavijo, Biol.2, Carlos Chacón, Microbiol.2, Salim Mattar, Ph.D.3
1. Joven Investigador, Universidad de Córdoba, Instituto de Investigaciones Biológicas del Trópico, Montería. Córdoba.
e-mail: jorgemirandaregino@gmail.com rodrigor333@yahoo.es
2. Investigador Corporación CorpoGen, Bogotá, Colombia. e-mail: ancla95@yahoo.com micanech@yahoo.co.uk
3. Profesor Titular, Director Instituto de Investigaciones Biológicas del Trópico, Universidad de Córdoba, Montería, Córdoba.
e:mail: mattarsalim@hotmail.com
Recibido para publicación mayo 10 de 2006 Aceptado para publicación septiembre 8, 2006
RESUMEN
Objetivo: Analizar la susceptibilidad antimicrobiana y la variabilidad genética de algunas cepas de Mycobacterium tuberculosis aisladas en la ciudad de Montería.
Métodos: Se incluyeron pacientes con diagnóstico clínico de tuberculosis y se tomaron datos sociodemográficos y epidemiológicos. Las muestras de esputos se cultivaron en el medio de Lowenstein-Jensen. Se llevaron a cabo las pruebas de susceptibilidad antimicrobiana por el método de las proporciones múltiples y rifoligotyping de segunda generación. La variabilidad genética se estudió a través del RFLP.
Resultados: Mycobacterium tuberculosis se aisló en 67 personas con tuberculosis pulmonar. La baciloscopia fue positiva en 59 (88%) y la edad estuvo entre 14 y 83 años (media 41.5 años). La resistencia fue 29.2% (14/48), y la multirresistencia 6.7% (3/48). La técnica rifoligotyping descubrió 4 cepas con mutación en el gen rpoB, región hot spot codón 531. El número de copias de IS6110 varió entre 3 y 13 y 12 (48%) cepas presentaron entre 8 y 9 copias. El RFLP permitió establecer dos grupos A y B. El grupo A estuvo conformado por 20 (80%) cepas, con un único cluster formado por los pacientes 17, 45, 54. El grupo B, conformado por 5 (20%) cepas, con un único cluster formado por los pacientes 31 y 32.
Conclusiones: Se demostró que en Montería, existe una alta tasa de resistencia a los fármacos anti-tuberculosos y una gran variabilidad genética de las cepas de M. tuberculosis. Se sugiere establecer medidas de vigilancia en epidemiología y salud pública para el control de esta enfermedad.
Palabras clave: Tuberculosis; Multirresistencia; Rifoligotyping; RFLP; Montería; Colombia.
Preliminary survey of antimicrobial susceptibility and genetic variability of Mycobacterium tuberculosis isolates from Colombian Caribbean
SUMMARY
Objective: The aim of this preliminary study was to investigate the drug susceptibility and genetic variability of Mycobacterium tuberculosis isolated from patients of Montería.
Methods: Patients with tuberculosis were included in the study. Socio-demographics and epidemiologic information was collected from each patient. Sputum samples were cultured on Lowenstein-Jensen medium. Antibiotic susceptibility testing was performed using the proportion method and rifoligotyping. The study of genetic variability was performed by RFLP.
Results: 67 strains of M. tuberculosis isolated from patients with pulmonary tuberculosis were included; fifty-nine patients (88%) were smear positive, the patients ranged from 14 to 83 of age, mean 41.5 years. The initial resistance and multidrug-resistance was 29.2% (14/48) and 6.7% (3/48) respectively. The rifoligotyping detected 4 strains (4, 17, 21 and 45) with the mutation in the rpoB gen, hot spot region codon 531. The number of IS6110-bands varied from 3 to 13, 12 (48%) isolates had among 8 and 9 copies. RFLP revealed genetic diversity of isolates in 2 groups: group A and B. Group A included 20 (80%) strains and only one cluster constituted by the patients 17, 45, 54. Group B, had 5 (20%) strains with only one cluster conformed by two patients 31 and 32.
Conclusions: The study demonstrated high levels of drug resistance and variability of strains of M. tuberculosis in Montería. Our results suggest establishing urgent measures of surveillance in epidemiology and public health for the control of the disease.
Keywords: Tuberculosis; Multidrug-resistance; Rifoligotyping; RFLP; Montería; Colombia.
Cada año cerca de 9 millones de personas se enferman de tuberculosis (TB) y mueren al menos 2 millones1. El resurgimiento de la tuberculosis a nivel mundial se debe sobre todo al creciente número de personas infectadas con el virus de la inmunodeficiencia humana (VIH) y al aumento en la frecuencia de cepas multirresistentes (MDR)2. En la actualidad la tuberculosis se concentra en subgrupos desprotegidos, tales como las poblaciones urbanas de menores recursos, los alcohólicos, los desplazados por el conflicto interno armado, usuarios de drogas intravenosas, las personas sin domicilio fijo y los internos de las penitenciarias. En la población de edad avanzada la enfermedad por lo general se debe a la reactivación de una infección adquirida en el pasado, mientras que en los niños de corta edad, adultos jóvenes y pacientes con el VIH la TB refleja una transmisión activa en la comunidad3.
En Colombia en el año 2002 la TB presentó una incidencia de 26 por 100,000 habitantes (11,376 casos); en el año 2003 se observó una leve disminución de 25.1 por 100,000 habitantes (11,217 casos)4. Para el año 2005 la tasa de incidencia de tuberculosis disminuyó en 22 casos por 100,000 habitantes (10,272 casos) observándose así una aparente disminución de los casos de TB a través de los años5. En el departamento de Córdoba, la incidencia de tuberculosis en los últimos años ha tenido un promedio de 34 casos por 100,000 habitantes. En la ciudad de Montería los casos de TB en los años 2000, 2001 y 2002 mantuvieron una incidencia promedio de 115 casos anuales, mientras que en 2003 y 2004 los casos aumentaron en 160 y 143 respectivamente (Comunicación, Secretaría de Salud Departamental de Córdoba, 2006).
Con respecto a la situación de la susceptibilidad antimicrobiana, en Colombia existen informes de resistencia global de 14.1% y multirresistencia de 1.8% del año 19925; en un segundo estudio la resistencia global fue de 15.6% y la multirresistencia 1.5% años 1999-20005,6. La resistencia de M. tuberculosis a los fármacos es atribuida a mutaciones genéticas que alteran los sitios de acción del antibiótico; esta resistencia es cromosómica, definitiva e irreversible7. Para descubrir las mutaciones se han desarrollado técnicas moleculares que permiten determinar con precisión mutaciones puntuales, inserciones o deleciones en los genes blanco de los antibióticos en M. tuberculosis, como por ejemplo la región de 81 pb localizada en el gen rpoB que codifica para subunidad beta de la ARN polimerasa. Esta región es conocida como región determinante de resistencia a rifampicina (RDRR) o región hot spot8. Las técnicas moleculares son más rápidas para la identificación de cepas resistentes de M. tuberculosis y en la actualidad se prefieren a la técnica de proporciones múltiples que se tarda más de un mes para obtener resultados9.
De otra parte, el entendimiento de la dinámica de la transmisión y la patogénesis de la tuberculosis ha avanzado con el desarrollo de métodos moleculares de tipificación10. El método más usado para la tipificación de M. tuberculosis es el RFLP (fragmentos de restricción de longitud polimórfica) que se basa en la variabilidad del número de copias y ubicación de la secuencia de inserción IS6110en el genoma bacteriano11. Esta variabilidad permite diferenciar o establecer un patrón de similaridad entre diferentes cepas de M. tuberculosis. También se usa para estudiar la diseminación de la TB en brotes y epidemias, analizar la transmisión, dinámica de la enfermedad y determinar los factores de riesgo de transmisión en una comunidad12-18. De igual modo juega un papel importante en la diferenciación entre reinfección exógena y reactivación endógena13,14,19, y también se puede utilizar en el laboratorio para identificar contaminación cruzada12,13,20,21.
En Montería no existen estudios en los últimos 50 años sobre la resistencia de TBC a las drogas antituberculosas de primera línea, ni cuál es la tasa de MDR. Tampoco se conocen estudios que muestren el polimorfismo genético de las cepas de M. tuberculosis y la existencia de clones comunes o diferentes que circulan en la ciudad. El objetivo de este trabajo preliminar es analizar la susceptibilidad antimicrobiana y la variabilidad genética de algunas cepas de M. tuberculosis aisladas en la ciudad de Montería.
MATERIALES Y MÉTODOS
Tipo de estudio. Estudio de tipo descriptivo prospectivo y realizado entre julio de 2003 y julio de 2005.
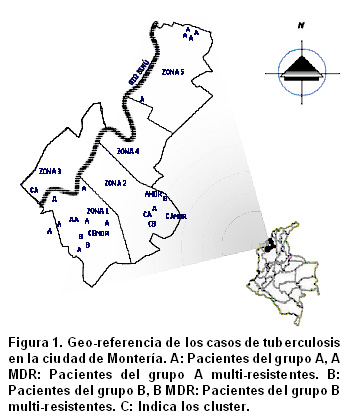
Área geográfica. Se localizó en la ciudad de Montería, capital del departamento de Córdoba con una población aproximada de 348,168 habitantes, situada a lo largo del río Sinú, con una temperatura promedio anual de 30ºC y una humedad de 90% (Figura 1).
De acuerdo con el plan de ordenamiento municipal y catastral, se establecieron 5 zonas geográficas: la zona 1 comprendió barrios del sur de la ciudad (estratos 1 y 2), zona 2 centro de la ciudad (estratos 1, 2 y 3), zona 3 barrios de la margen izquierda del río Sinú (estratos 1 y 2), zona 4 área norte (estratos 4 y 5) y zona 5 otros barrios del área norte (estratos 1 y 2), (Figura 1).
Población de estudio. La prevalencia anual de casos de TB en Montería es de 140 casos (Comunicación, Secretaría de Salud Departamental de Córdoba, 2006). Se calculó que se podrían obtener 20 (14%) pacientes anuales de esta prevalencia lo que daría un número de 60 cepas estudiadas en tres años. Sin embargo, por problemas presupuestales se estudiaron sólo 48 (11% anual) para susceptibilidad y 35 (25%) para RFLP. Con estos datos se asumió que la población escogida representaría un porcentaje importante de la prevalencia de las infecciones por TB en la ciudad.
A los pacientes se les tomaron datos sociodemográficos y epidemiológicos, como edad, género, procedencia, estado serológico para VIH, fecha de diagnóstico, tratamiento previo y contactos con enfermos tuberculosos. Los datos fueron suministrados por el personal de salud del Programa de Tuberculosis del municipio de Montería. No se obtuvo información acerca del uso de drogas, consumo de alcohol o antecedentes de fumador; 60 (90%) eran casos nuevos y 7 (10%) de ellos habían sido previamente tratados con medicamentos antituberculosos.
Criterios de inclusión y exclusión. Se incluyeron personas con diagnóstico clínico y radiológico y a quienes se les aisló M. tuberculosis en el cultivo. Como criterio de exclusión se tomaron a personas sin baciloscopia positiva, sin radiografía de tórax sugestiva de TB y con más de 2 meses de tratamiento con medicamentos antituberculosos.
Especímenes. Los especímenes fueron esputos y se procesaron en el Instituto de Investigaciones Biológicas del Trópico de la Universidad de Córdoba. Las muestras fueron digeridas y decontaminadas por el método Ticket y Ticqson22. La tinción de los esputos se llevó a cabo con la coloración de Kinyoun. El cultivo se realizó en el medio Lowenstein Jensen y se incubaron a 37ºC durante seis semanas.
Prueba de susceptibilidad a medicamentos antituberculosos. El método utilizado para la determinación de la resistencia a isoniacida (INH), rifampicina (RIF), etambutol (EMB), estreptomicina (SM) y tioacetazona (TIO) fue el de las proporciones múltiples de Canetti, Rist y Grosset23. Las concentraciones fueron las siguientes: INH: 0.2 µg/ml, RIF: 40 µg/ml, EMB 2 µg/ml, STR 4 µg/ml, TIO 2 µg/ml. Las pruebas de susceptibilidad antimicrobianas se realizaron en el Laboratorio de Salud Pública Departamental del Atlántico. Por problemas presupuestales no se hizo la prueba de susceptibilidad a todos los aislados. Los resultados de resistencia se expresaron en porcentaje para cada medicamento y se dividieron en dos grupos según los criterios actuales de clasificación: resistencia en casos nuevos y casos previamente tratados24.
Un microorganismo se consideró como resistente cuando mostró por lo menos resistencia a un medicamento de los cuatro estudiados; la multirresistencia fue definida como resistencia a INH y RIF con o sin resistencia a otros fármacos2. De los 67 gérmenes aislados sólo se incluyeron 48 para ser analizados por el método de las proporciones múltiples. Estos aislados se escogieron al azar a través de una tabla de números aleatorios impares, con excepción del paciente recluido en la prisión que se incluyó en el trabajo.
Análisis por rifoligotyping. Para determinar la resistencia a nivel genotípico se utilizó rifoligotyping de segunda generación25. Cada región del gen rpoB fue amplificada con los primers rpo-d (5’ CGC CGC GAT CAA GGA GT 3’) y Biorpo-r (5´ACG TCG CGG ACC TCC A 3’), e hibridados contra 13 sondas específicas correspondientes a 6 que identificaban los genotipos rifampicino-susceptibles y 7 de las secuencias mutantes más comunes en la región hot spot del gen rpoB. Como control de amplificación, se incluyeron en el rifoligotyping controles positivos para las mutaciones 516(GTC), 516(TAC), 526(TAC), 526(GAC), 529(CAA), 531(TGG) y 531(TTG), un ADN de la cepa H37Rv como control silvestre y ADN de M. smegmatis y M. bovis pertenecientes al complejo y por tanto con hibridación positiva como control de especificidad. Los productos hibridados se visualizaron con AP-SA-CDP® Star system (KPL inc, Maryland, USA).
Análisis de los perfiles genéticos. A partir de un cultivo de M. tuberculosis en caldo 7H9 se extrajo el ADN siguiendo el protocolo descrito por Cardoso et al.26 La lisis se llevó a cabo con lisozima, proteinasa K y SDS 1% (P/V). El ADN se extrajo con CTAB 10% (cetyltrimethylammoniumbromide). El análisis de southern blot se llevó a cabo siguiendo el protocolo descrito por Sambrok et al.27 El ADN se digirió con la enzima de restricción Pvull y los fragmentos se separaron en gel de agarosa al 0.8% teñido con bromuro de etidio. El ADN fue transferido a una membrana de nylon Millipore® por capilaridad y se utilizó una sonda de 245 pb (pGEM245 INS) que hibrida con uno de los brazos de IS6110. El descubrimiento se llevó a cabo con el estuche de quimioluminiscencia de fosfatasa alcalina de KPL (Maryland, USA) siguiendo las indicaciones del fabricante. Como control externo se incluyó una cepa de referencia de M. tuberculosis H37Rv. Para estudiar la diversidad clonal de los aislamientos se generó un dendrograma de similitud basado en el algoritmo UPGMA (Nei 1972)28 y el coeficiente de Dice. Se definió como cluster si dos o más aislamientos presentaba idéntico patrón RFLP-IS6110. De las 67 cepas aisladas, sólo se analizaron 35 por RFLP, las cuales se escogieron aleatoriamente utilizando una tabla de números impares. Por problemas presupuestales no se tipificaron los 67 aislamientos.
La amplificación por PCR de la secuencia de inserción IS6110, se llevó a cabo de acuerdo con Liébana et al.29 Se utilizaron los iniciadores IS41 (5’ (IS41, 5’ CCT GCG AGC GTA GGC GTC GG 3’) e IS43 (5’ TCA GCC GCG TCC ACG CCG CCA 3’). Se tomaron 0.05 unidades de Taq Polimerasa (TucanTaq CorpoGen), 10pmoles de cada iniciador, 1.5 mM de MgCl2, 0.2 mM de deoxinucleotidos y 50 ng de ADN en un volumen final de 25 µl. La PCR se llevó a cabo en un termociclador MJ Reasearch PT100, usando un ciclo de 94ºC por 4 minutos, 30 ciclos de denaturación a 94ºC por un minuto, anillaje a 68ºC por un minuto, extensión a 72ºC por 75 segundos, y una extensión final a 72ºC por 4 minutos.
Aspectos éticos . Se siguieron las normas técnicas, científicas y administrativas para la investigación en salud del Ministerio de Salud de Colombia, Resolución N° 008430 de octubre 4 de 1993. A lo largo del estudio siempre se protegió la privacidad e intimidad del paciente, con con un número interno de identificación. Los resultados de los análisis se incluyeron en una base de datos para usarlos de forma anónima mediante la asignación de un número. A todos los sujetos ingresados se les explicó verbalmente el tipo de estudio y se obtuvo su consentimiento oral y escrito. La investigación fue aprobaba por el Comité de Ética del Instituto de Investigaciones Biológicas del Trópico de la Universidad de Córdoba, Montería
Procesamiento y análisis de datos. Los datos se recolectaron mediante el uso de un formulario estandarizado, se integraron a una base de datos y se procesaron y analizaron utilizando los recursos y análisis de datos del programa de Excel® 2000.
RESULTADOS
Características clínicas y sociodemográficas. La edad de los pacientes estuvo entre el rango de 14 a 83 años, con una media de 41.5 años. La distribución por edad fue la siguiente: 14 (21%) pacientes entre 14 y 24 años; 28 (42%) entre 25 y 45 años; 18 (27%) entre 46 y 64 años; y 7 (10%) pacientes mayores de 65 años. De los 67 pacientes, 38 (56%) eran de sexo masculino. Todos fueron diagnosticados con tuberculosis pulmonar; la baciloscopia fue positiva para 59 (88%) pacientes; los 8 restantes fueron diagnosticados por radiología sugestiva de TB y por cultivo. Se encontró evidencia de historia previa de TB en 7 de ellos y 2 tuvieron diagnóstico positivo para VIH; 45 (67%) de los pacientes había sido vacunado con la BCG (cicatriz BCG) y 22 (33%) no eran vacunados. Los individuos sin baciloscopia se diagnosticaron por criterios clínicos y radiológicos sugestivos de TB.
La distribución de los pacientes en las 5 zonas de la ciudad fue así: 34 (51%) estuvieron ubicados en la zona 1; 12 (18%) en la zona 2; 7 (10%) en la zona 3; 13 (20%) en la zona 5; un paciente no tenía hogar y no se presentó ningún caso en la zona 4.
Susceptibilidad antimicrobiana. De las 48 cepas de M. tuberculosis analizadas, 41 (85.5%) pertenecían a pacientes nuevos y 7 (14.5%) a casos previamente tratados. La resistencia global fue 29.2% (14/48) y la multirresistencia 6.7% (3/48).
La resistencia frente a cada uno de los medicamentos en pacientes nuevos fue: 7 (17%) cepas resistentes a INH, seguida de 4 (9.7%) resistentes a SM y 3 (7.3%) a EMB. Ninguna cepa en los pacientes nuevos presentó resistencia a RIF y ninguno tuvo cepas MDR. Otras asociaciones no MDR en pacientes nuevos fueron INH+SM en 2; otras combinaciones de resistencia se muestran en el Cuadro 1.
En las personas previamente tratadas se encontró resistencia en 4 (57%) cepas y 3 (43%) sensibles. Ninguna cepa presentó resistencia a un solo fármaco; se observaron 3 cepas MDR, una de ellas resistente a INH+RMP+ EMB+SM que se aisló de una persona recluida en un centro carcelario. Otras asociaciones no MDR encontradas fueron INH+SM y INH+EMB (Cuadro 1). Ninguna cepa presentó resistencia a tioacetazona.
Rifoligotyping. Se encontraron mutaciones en el gen rpoB en las cepas 4, 17, 21 y 45, todas estas en la región hot spot codón 531, con sustitución TCG (Ser) por TTG (Leu) (Figura 2). La cepa 45 mostró discrepancia en los resultados, pues fue sensible por el método de las proporciones múltiples y resistente por rifoligotyping; las cepas 4, 17, 21 que presentaron mutación en el gen rpoB eran MDR.
Análisis de los perfiles genéticos. El número de copias de IS6110 en todas las cepas varió entre 3 y 13; 12 (48%) cepas presentaron entre 8 y 9 copias; 5 (20%) tuvieron 6 copias; 5 (20%) presentaron entre 10 y 13 copias; 2 (8%) cepas presentaron 7 copias; y sólo una cepa presentó 3 (4%) copias. Diez de las 35 cepas analizadas no presentaron ningún perfil genético y no poseían la secuencia de inserción IS6110 porque se comprobó su ausencia.
A través del coeficiente de similaridad de 70%, se establecieron los grupos A y B. El grupo A estuvo conformado por 20 (80%) (Figura 3), con un único cluster formado por los pacientes 17, 45, 54; los pacientes 17 y 45 residían en una misma zona y en el análisis molecular de la resistencia presentaron mutación en el gen rpoB. Sin embargo, sus patrones de susceptibilidad con respecto a otros medicamentos fueron diferentes (Cuadro 2).
Las cepas 40, 90, 91 y 93 del grupo A estuvieron relacionadas genéticamente con 94% de similaridad; estas personas residían en diferentes zonas de la ciudad. Tres de estos, 90, 91 y 93, se diagnosticaron entre junio y julio del año 2005, mientras que el paciente 40 se diagnosticó en abril de 2004.
Los pacientes 25 y 26 tuvieron 95% de similaridad; residían en zonas diferentes de la ciudad y se diagnosticaron entre julio y noviembre de 2003. Los pacientes 21 y 23, al igual que los dos pacientes anteriores, tuvieron un porcentaje de similaridad de 95%. Al revisar la historia clínica se encontró que el paciente 21 tenía historia previa de TB y se encontraba recluido en una institución penitenciaria de la cuidad y presentó resistencia a 4 de los fármacos de la terapia antituberculosa y se encontró mutación en el gen rpoB; mientras que el paciente 23 sólo tuvo resistencia a INH y no presentó mutación en el gen rpoB. Ambos pacientes fueron diagnosticados entre julio y septiembre de 2003.
El grupo B, estuvo conformado por 5 20%) cepas, con un único cluster formado por dos pacientes (31 y 32). Al revisar la historia clínica se encontró que el paciente 31 tenía historia previa de TB; ambos presentaron resistencia a isoniazida y estreptomicina pero no presentaban relación epidemiológica alguna (Cuadro 2). Los otros 3 pacientes (4 TBC reincidente, 59 y 60 con porcentaje de similaridad de 81%), tampoco estuvieron relacionados epidemiológicamente.
En total 22 perfiles genéticos RFLP-IS6110 se obtuvieron de los 25 aislamientos. Cinco (20%) perfiles genéticos se agruparon en dos clusters y 20 perfiles RFLP-IS6110 tuvieron ocurrencia única. La (Figura 1) muestra el mapa de la ciudad de Montería, con la estratificación de las diferentes zonas incluidas en el estudio y la geo-referencia de los pacientes en los clusters A y B y las cepas MDR.
DISCUSIÓN
La tuberculosis constituye unos de los principales problemas de salud pública en el departamento de Córdoba con una incidencia de 25.5 por 100,000 habitantes en el año 2005 y Montería, con un promedio anual de 129 casos por 100,000 habitantes durante los últimos 5 años (2000-2005) han sido considerados como territorios de riesgo medio para TB.
De otro lado, muy a pesar de que los departamentos poseen recursos extras del Plan de Atención Básica, en Colombia el nuevo Sistema de Seguridad Social ha permitido el aumento de la TB, porque se descentralizaron los programas verticales de control que en un principio se manejaban desde el nivel central del Estado. Como consecuencia, se redujo la captación de sintomáticos respiratorios, estudios de contactos y la adherencia de pacientes con TB a los esquemas de medicación; también se redujo la inversión de los recursos para prevención en las entidades estatales, municipales y departamentales, las EPS y las ARS, afectando así los procesos de control de enfermedades tan importantes como la TBC30.
En el presente estudio todos los pacientes se ubicaron en los estratos 1 y 2 y 90% de ellos estuvo en el grupo de edad con mayor productividad económica (entre 15 y 54 años) lo que confirma que la tuberculosis afecta sobre todo a las poblaciones más desprotegidas y que en los países en vías de desarrollo, más de 75% de los casos diagnosticados con TB se encuentra en el rango de edad de mayor productividad económica31.
La multirresistencia es un fenómeno global; existen regiones en el mundo con una alta incidencia de cepas MDR como las encontradas en Estonia (14.1%), la provincia de Henan en la China (10.8%), Letonia (9%), y las provincias rusas de Ivanovo (9%)32. La multirresistencia encontrada en este artículo fue 6.7% similar a la informada en Buenaventura 6% por Moreira et al.32 Estas cifras de multirresistencia son altas comparadas con las del promedio nacional 1.5% y con el promedio global de 1%6. Sin embargo, la MDR obtenida del último estudio nacional de resistencia fue 9.6%, similar a las de regiones con alta incidencia de cepas MDR en el mundo. Es importante recalcar que la MDR encontrada en este estudio (6.7%) correspondió a pacientes previamente tratados, que presentan las más altas tasas de MDR en Colombia5.
La resistencia global del presente estudio (29.2%) también es elevada si se compara con el último estudio nacional de vigilancia de la resistencia de M. tuberculosis en donde informan cifras de resistencia global de 20.7%5. Ninguna cepa aislada de pacientes no tratados presentó MDR. En Colombia la MDR en personas no tratadas se ha mantenido en niveles estables al comparar los estudios realizados entre 1999 y 2000 con los efectuados en los años 2004 y 2005 por el Intituto Nacional de Salud5.
La alta frecuencia de resistencia es un indicador importante del insuficiente control en los programas antituberculosos de una determinada región. Las personas infectados con VIH y con cepas de M. tuberculosis resistentes a múltiples medicamentos son difíciles de curar y el tratamiento alternativo es más tóxico y costoso33.
El caso del enfermo con TB que se encontraba recluido en el centro penitenciario, merece especial atención, porque las cárceles conforman un sistema cerrado que acoge a una población que por sus características sociales, condiciones de hacinamiento, hábitos tóxicos y elevada incidencia de infección por VIH, son reconocidas como un lugar de alto riesgo para la transmisión de la TB34. En este caso el recluso convivía con otras personas que posiblemente pudieron infectarse con esta cepa MDR. Los programas de prevención y control de la TB en las cárceles deben ser rápidos en la localización de los pacientes con la enfermedad, en estudios de contactos y en el tratamiento estrictamente supervisado. Al paciente recluido en el centro penitenciario y sus contactos no se le realizó seguimiento.
La resistencia a RIF en M. tuberculosis está a menudo asociada con resistencia a isoniacida y a otros fármacos antituberculosos. Esto ha sugerido que la determinación de la resistencia a RIF juega un papel clave en la vigilancia de la TB MDR35,36. En el presente estudio se encontraron 4 cepas que tenían mutación en el gen rpoB; de éstas, sólo 3 (75%) cepas presentaron resistencia a isoniazida (cepas MDR). Las 4 cepas tenían la misma mutación en la región hot spot codón 531, mutación tipo sustitución de TCG (ser) por TTG (Leu). Se ha informado un elevado porcentaje de cepas de M. tuberculosis resistentes a RIF que explican su resistencia por mutaciones puntuales, pero también por inserciones o deleciones dentro de la región determinante de resistencia a RIF ubicada entre los codones 511 a 533 del gen rpoB 37. Como se observó previamente, los hallazgos de este artículo muestran que todos los aislados resistentes a rifampicina tenían la misma mutación la cual es común en Colombia y en otros países6,36-41.
El resultado discrepante en la cepa 45 por rifoligotyping y el método de las proporciones múltiples, pudo deberse a la alta sensibilidad de la PCR capaz de amplificar un segmento de la población resistente y no revelada por el método de las proporciones. Porras et al.6 y Cohen et al.42 informaron hallazgos similares en Colombia.
Comparado con el método de las proporciones múltiples y en general con los métodos fenotípicos de descubrimiento de multirresistencia, que requieren aproximadamente entre 28 y 42 días para arrojar un resultado, la rapidez en la detección de MDR es la principal ventaja de los métodos moleculares36.
De otro lado, las técnicas de biología molecular aplicadas al estudio de la TB han permitido comprender la dinámica de la transmisión, la patogenicidad y los factores de riesgo asociados con la enfermedad. En este estudio se usó la aplicación de la técnica molecular RFLP para determinar variabilidad genética de las cepas de M. tuberculosis y la existencia de clones comunes o diferentes que circulan en la ciudad.
Sólo se tipificaron 35 de las 67 cepas, donde 10 no presentaron patrón RFLP, así que sólo se analizaron 25. Con respecto a las diez cepas que no presentaron ningún perfil genético, probablemente estas no poseían la secuencia de inserción IS6110 porque se comprobó la ausencia de la IS6110 por PCR. En ese sentido, varios autores han informado el aislamiento de cepas de M. tuberculosis sin la secuencia de inserción IS611013,16,43. Se han obtenido cepas con pocas copias o ausencia de IS6110 pero que presentan un mejor polimorfismo cuando son tipificadas con técnicas como spoligotyping (direct repeat locus) asociado con VNTR (variable number tandem repeat) o MIRU (mycobacterial interspersed repetitive units). Estos métodos nuevos de tipificación demuestran ser útiles en la epidemiología molecular de M. tuberculosis y se consideran como alternativos al tradicional RFLP-IS611044.
En los 25 aislamientos que sí presentaron patrón de RFLP, el mayor porcentaje de copias encontradas estuvo entre 8 y 9 copias de IS6110 (48%); este hallazgo concuerda con otros estudios realizados en Colombia por Gómez et al.43, quien informó 9 copias de IS6110 como el perfil más común en cepas aisladas en diferentes parte del país. En el presente estudio, sólo una cepa presentó 3 (4%) copias; este porcentaje bajo de cepas con pocas copias de IS6110 es similar al obtenido en Cuba por Díaz et al.45y Baldiviano et al.46 en Perú, quienes informaron respectivamente entre 4.1% y 5.4% de cepas con bajo número de copias de IS6110. Las cepas con bajo número de copias encontradas en la región de las Américas difieren de las encontradas en Vietnam, Isla de Guadalupe y Tanzania donde es más frecuente encontrar pocas copias de IS611043. Esto soporta la hipótesis de que ciertos genotipos específicos de M. tuberculosis están asociados con regiones geográficas específicas45.
En este estudio preliminar las cepas que no presentaron patrón RFLP, no se analizaron por otro método de tipificación como spoligotyping; este último podría facilitar en un futuro un mejor entendimiento de la dinámica poblacional de M. tuberculosis en Montería.
El porcentaje de clusters en una determinada población de pacientes se usa para indicar el grado de transmisión reciente de la TB. Si bien no fue uno de los objetivos del estudio establecer la transmisión activa por el porcentaje de agrupamiento, los autores encontraron que 20% de los enfermos estaban agrupados, indicando transmisión reciente en la comunidad. Sin embargo, se debe tener en cuenta, como una limitación de este estudio que el número de pacientes analizados por RFLP es bajo y no refleja el valor real del grado de agrupamiento de las cepas de M. tuberculosis en la ciudad. Otros informes demuestran que una reducida muestra subestima enormemente el agrupamiento47,48.
Aunque un cluster de aislamientos bacterianos puede poseer perfiles idénticos con 100% de similitud, se han obtenido pacientes con relación epidemiológica estrecha y comprobada que pueden presentar diferencias en sus perfiles genéticos, como por ejemplo ausencia o presencia de una o varias bandas adicionales46,49.
La investigación epidemiológica realizada durante el período de recolección de muestras, no mostró relación directa entre los pacientes. Después de obtener los resultados del RFLP, se investigó con detalle a las personas que estuvieron en un mismo cluster. Los datos demostraron que los individuos no tenían relación directa con otros del mismo cluster. Además, estos tampoco mostraron relación con la zona geográfica donde residían. Es posible que estos pacientes con idéntico perfil genético adquirieran la infección de un caso previo que no fue incluido en el estudio y que estuviera relacionado epidemiológicamente con los pacientes de un cluster50. Otros estudios han revelado que individuos no relacionados epidemiológicamente pueden presentar el mismo patrón de RFLP, debido a que las cepas pueden ser transmitidas durante un leve contacto entre las personas sin aparente relación epidemiológica51,52.
En conclusión, los hallazgos previos de este estudio, muestran 22 perfiles diferentes indicando una alta variabilidad genética de las cepas de M. tuberculosis estudiadas. El análisis por RFLP mostró algunas cepas con idéntico perfil genético y diferente patrón de susceptibilidad antimicrobiana, lo que sugiere que la resistencia pudo ser adquirida por un mal manejo terapéutico.
Este estudio preliminar permitió demostrar que en la ciudad de Montería, existe una tasa alta de resistencia a los fármacos anti-tuberculosos que sugieren establecer medidas urgentes de vigilancia epidemiológica y salud pública para el control de esta enfermedad.
AGRADECIMIENTOS
A Colciencias y la Universidad de Córdoba por su apoyo al programa de jóvenes investigadores convenio interadministrativo especial de cooperación N° 089-2004. Al Laboratorio de Salud Pública del Atlántico, Sección de Micobacterias; a la Secretaría de Salud y Seguridad Social Municipal de Montería y al Laboratorio Departamental de Salud Pública.
REFERENCIAS
1. Baltussen R, Floyd K, Dye C. Cost effectiveness analysis of strategies for tuberculosis control in developing countries. BMJ 2005; 331: 1364-1369.
2. World Health Organization. Anti-tuberculosis drug resistance. World Report N° 3, 2004. WHO, 2005.
3. Haas D, Des Prez R. Enfermedades por micobacterias: Mycobacterium tuberculosis. In: Mandell & Benett (eds.). Enfermedades infecciosas]. 4ª ed. New York: Churchill Livingstone; 1996. p. 2480-2513.
4. Instituto Nacional de Salud de Colombia. Día Mundial de la Tuberculosis 2004. [Fecha de acceso enero 26 de 2005]. URL disponible en: http://www.ins.gov.co/pdf_investiga/2004_tbc_dm.pdf
5. Instituto Nacional de Salud. Vigilancia de la resistencia de Mycobacterium tuberculosis a los medicamentos. Colombia 2004-2005. [Fecha de acceso agosto 14 de 2006]. URL disponible en: http://www.ins.gov.co/pdf/rnl/informe_final_EVR_2004_2005.pdf
6. Porras TB, León CI, Guerrero MI, Martin A, Portaels F, Palomino JC. Evaluación de métodos fenotípicos y genotípicos para la detección de farmacorresistencia de Mycobacterium tuberculosis. Biomedica 2005; 25: 22-33.
7. Tobón AM. Tratamiento de la tuberculosis multirresistente. Infectio 2001; 5: 260-265.
8. Jenkins C, Claxton AP, Shorten RJ, McHugh TD, Gillespie SH. Rifampicin resistance in tuberculosis outbreak, London, England. Emerg Infect Dis 2005; 11: 931-934.
9. Kremer K, Van Zetten M, Van Embden J, Schouls L, Van Soolingen D. «RIFO ASSAY» a PCR + reverse line blot hybridization method to detect rifampin resistance. Bilthoven: National Institute of Public Health and Environment; 2003.
10. Tazi L, El Baghdadi J, Lesjean S, Locht C, Supply P, Tibayrenc M, et al. Genetic diversity and population structure of Mycobacterium tuberculosis in Casablanca, a Moroccan city with high incidence of tuberculosis. J Clin Microbiol 2004; 42: 461-466.
11. Burger M, Raskin S, Brockkelt S, Amthor B, Geiss HK, Haas WH. DNA fingerprinting of Mycobacterium tuberculosis complex culture isolates collected in Brazil and spotted onto filter paper. J Clin Microbiol 1998; 36: 573-576.
12. van Soolingen D, Borgdorff MW, de Haas PEW, Sebek MM, Veen J, Dessens M, et al. Molecular Epidemiology of Tuberculosis in the Netherlands: a Nationwide Study from1993 through 1997. J Infect Dis 1999; 180: 726-736.
13. Das SD, Narayanan S, Hari L, Hoti SL, Thangathurai RK, Charles N, et al. Differentiation of highly prevalent IS6110 single-copy strains of Mycobacterium tuberculosis from a rural community in South India with an ongoing DOTS programme. Infect Genet Evol 2005; 5: 67-77.
14. Narayanan S. Molecular epidemiology of tuberculosis. Indian J Med Res 2004; 120: 233-247.
15. Kiers A, Drost AP, van Soolingen D, Veen J. Use of DNA fingerprinting in international source case finding during a large outbreak of tuberculosis in The Netherlands. Int J Tuberc Lung Dis 1997; 1: 239-245.
16. Moonan PK, Bayona M, Quitugua TN, Oppong J, Dunbar D, Jost JrKC, et al. Using GIS technology to identify areas of tuberculosis transmission and incidence. Int J health Geogr 2004; 3: 23-32.
17. Chin DP, Crane CM, Diul MY, Sun SJ, Agraz R, Taylor S, et al. Spread of Mycobacterium tuberculosis in a community implementing recommended elements of tuberculosis control. JAMA 2000; 283: 2968-2974.
18. van Deutekom H, Gerritsen JJ, van Soolingen D, van Ameijden EJ, van Embden JD, Coutinho RA. A molecular epidemiological approach to studying the transmission of tuberculosis in Amsterdam. Clin Infect Dis 1997; 25: 1071-1077.
19. Small PM, Shafer RW, Hopewell PC, Singh SP, Murphy MJ, Desmond E, et al. Exogenous reinfection with multidrug-resistant Mycobacterium tuberculosis in patients with advanced HIV infection. N Engl J Med 1993; 328: 1137-1144.
20. Bauer J, Thomsen VO, Poulsen S, Andersen AB. False-positive results from cultures of Mycobacterium tuberculosis due to laboratory cross-contamination confirmed by restriction fragment length polymorphism. J Clin Microbiol 1997; 35: 988-991.
21. Cohn DL, O’Brien RJ. The use of restriction fragment length polymorphism (RFLP) analysis for epidemiological studies of tuberculosis in developing countries. Int J Tuberc Lung Dis 1998; 2: 16-26.
22. Tison F, Devulder B. Current methods of isolation and identification of micobacteria. Pathol Biol 1965; 13: 458-462.
23. Canetti G, Fox W, Khomenko A, Mahler N, Menon NK, Mitchinson DA, et al. Advances in the techniques of testing mycobacterial drug sensitivity and the use of sensitivity test in tuberculosis control programs. Bull World Health Organ 1969; 41: 21-43.
24. Montoro E, Echemendia M, Lemus D, Marrero A, Llanes MJ, Valdivia JA. Vigilancia de la resistencia de Mycobacterium tuberculosis a las drogas antituberculosas en Cuba, 1995-1998. Biomedica 2004; 24 (Supl): 80-84.
25. Rossau R, Traore H, De Beenhouwer H, Mijs W, Jannes G, De Rijk P, et al. Evaluation of the INNO-LiPA Rif. TB assay, a reverse hybridization assay for the simultaneous detection of Mycobacterium tuberculosis complex and its resistance to rifampin. Antimicrob Agents Chemother 1997; 41: 2093-2098.
26. Cardoso-Leão S, Martin A, Mejía G, Palomino J, Robledo J, Telles M, et al. Molecular protocols. Practical handbook for phenotypic and genotypic identification of Mycobacteria. Brussels: European Commision, International Co-operation for Developing Countries; 2004. p. 113-125.
27. Sambrook J, Fritsch E, Maniatis T. Analysis and cloning of eucaryotic genomic DNA. In: Nolan N (ed.). Molecular cloning, a laboratory manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press; 1998. p. 9-62.
28. Nei M. Genetic distance between populations. Am Nat 1972; 106: 283-292.
29. Liébana E, Aranaz A, Mateos A, Vilafranca M, Gómez-Mampaso E, Tercero JC, et al. Simple and rapid detection of Mycobacterium tuberculosis complex organisms in bovine tissue samples by PCR. J Clin Microbiol 1995; 33: 33-36.
30. Carvajal R, Cabrera GA, Mosquera J. Percepciones de los efectos de la implementación del Sistema General de Seguridad Social en Salud sobre las acciones de control de tuberculosis en el Valle del Cauca, Colombia. Colomb Med 2004; 35: 179-184.
31. Machado JE, Martínez JW. Asociación tuberculosis y VIH en pacientes de Pereira, Colombia. Colomb Med 2005; 36: 239-243.
32. Moreira CA, Hernández HL, Arias NL, Castaño MC, Ferro BE, Jaramillo E. Resistencia inicial a drogas antituberculosas en Buenaventura, Colombia. Biomedica 2004; 24: 73-79.
33. Laxminarayan R, Bhutta Z, Duse A, Jenkins P, O’Brien T, Okeke IN, et al. Drug resistance. In: Jamison DT, Breman JG, Measham AR, Alleyne G, Claeson M, Evans DB, et al. (eds.) Disease control priorities in developing countries. 2nd ed. Washington, DC: IBRD/The World Bank and Oxford University Press; 2006. p. 1031-1052.
34. Fernández JI, Fernández K, Catalán S, Alonso M, Chavese F. Transmisión de la tuberculosis en las prisiones de Madrid. Med Clin 2000; 115: 246-250.
35. Drobniewski F, Wilson S. The rapid diagnosis of isoniazida and rifampicin resistance in Mycobacterium tuberculosis a molecular story. J Med Microbiol 1998; 47: 189-196.
36. Guerrero MI, Suffys PN, Vanderborcht B, De Oliveira MM, Cohen IB, León CI. Comparación de métodos moleculares útiles en la detección rápida de Mycobacterium tuberculosis multirresistente (MTB-MDR). Infectio 2001; 5: 203-212.
37. Morris S,Bai GH, Suffys P, Portillo-Gómez L, Fairchok M, Rouse D. Molecular mechanisms of multiple drug resistance in Clinical isolates of Mycobacterium tuberculosis. J Infect Dis 1995; 171: 954-960.
38. Becerra F, Galindo A, Patiño M, Sánchez L, Lorente A, Del Portillo P, et al. Secuenciación de un fragmento amplificado del gen rpoB en cepas de Mycobacterium tuberculosis resistentes a rifampicina. Medicas UIS 2003; 17: 107-111.
39. Viveiros M, Leandro C, Rodrigues L, Almeida J, Bettencourt R, Couto I, et al. Direct application of the INNO-LiPA Rif.TB line-probe assay for rapid identification of Mycobacterium tuberculosis complex strains and detection of rifampin resistance in 360 smear-positive respiratory specimens from an area of high incidence of multidrug-resistant tuberculosis. J Clin Microbiol 2005; 43: 4880-4884.
40. Samper S, Iglesias MJ, Rabanaque MJ, Gómez LI, Lafoz MC, Jiménez MS, et al. Systematic molecular characterization of multridrug-resistant Mycobacterium tuberculosis complex isolates from Spain. J Clin Microbiol 2005; 43: 1220-1227.
41. Morcillo N, Zumarraga M, Alito A, Dolmann A, Schouls L, Cataldi A, et al. A low cost, home-made, reverse-line blot hybridization assay for rapid detection of rifampicin resistance in Mycobacterium tuberculosis. Int J Tuberc Lung Dis 2002; 6: 959-965.
42. Cohen IB. Caracterización de eventos en la resistencia a rifampicina de cepas colombianas de Mycobacterium tuberculosis. Bogotá: Universidad Nacional de Colombia; (Tesis Maestría) 2002.
43. Gómez-Marín JE, León CI, Inirida MG, Rigouts L, Portaels F. IS6110 Fingerprinting of sensitive and resistant strains (1991-1992) of Mycobacterium tuberculosis in Colombia. Mem Inst Oswaldo Cruz 2002; 97:1-4.
44. Brudey K, Driscoll JR, Rigouts L, Prodinger WM, Gori A, Al-Hajoj SA, et al. Mycobacterium tuberculosis complex genetic diversity: mining the fourth international spoligotyping database (SpolDB4) for classification, population genetics and epidemiology. BMC Microbiol 2006; 6: 23.
45. Díaz R, Gómez RI, Restrepo E, Rumbaut R, Sevy-Couyt J, Valdivia JA, et al. Transmission of tuberculosis in Havana, Cuba: a molecular epidemiological study by IS6110 restriction fragment length polymorphism typing. Mem Inst Oswaldo Cruz 2001; 96: 437-443.
46. Baldeviano C, Quispe N, Bonilla C, Gastiaburu D, Pro J, Llanos-Zavalaga L. Perfiles genéticos (RFLP-IS6110) y resistencia a drogas en aislamientos de M. tuberculosis de pacientes internados en un hospital referencial del Callao, Perú. Rev Peru Med Exp Salud Publica 2003; 20: 72-77.
47. Murray M. Sampling bias in the molecular epidemiology of tuberculosis. Emerg Infect Dis 2002; 8: 363-369.
48. Glynn JR, Vynnycky E, Fine PEM. Influence of sampling on estimates of clustering and recent transmission of Mycobacterium tuberculosis derived from DNA fingerprinting techniques. Am J Epidemiol 1999; 149: 366-371.
49. Alito A, Morcillo N, Scipioni S, Dolmann A, Romano MI, Cataldi A, et al. The IS6110 restriction fragment length polymorphism in particular multidrug-resistant Mycobacterium tuberculosis strains may evolve too fast for reliable use in outbreak investigation. J Clin Microbiol 1999; 37: 788-791.
50. Durmaz R, Ozerol HI, Durmaz B, Gunal S, Senoglu A, Evliyaoglu E. Primary drug resistance and molecular epidemiology of Mycobacterium tuberculosis isolates from patients in a population with high tuberculosis incidence in Turkey. Microbial Drug Resistance 2003; 4: 361-366.
51. Small PM, Hopewell PC, Singh SP, Paz A, Parsonnet J, Ruston DC, et al. The epidemiology of tuberculosis in San Francisco a population-based study using conventional and molecular methods. N Engl J Med 1994; 330: 1703-1709.
52. Tabet SR, Goldbaum GM, Hoonton TM, Eisenach KD, Cave MD, Nolan CM, et al. Restriction fragment length polymorphism analysis detecting a community-based tuberculosis outbreak among persons infected with human immunodeficiency virus. J Infect Dis 1994; 169: 189-192.