Diversidad genética en poblaciones humanas de dos regiones colombianas
Fernando Rondón, Cand PhD1, Julio César Osorio, BSc2, Ángela Viviana Peña, BSc2, Héctor Andrés Garcés, BSc2, Guillermo Barreto, MSc, PhD3
1. Estudiante de doctorado en Ciencias-Biología, Departamento de Biología, Facultad de Ciencias Exactas y Naturales, Universidad del Valle, Cali, Colombia. e-mail: ferchogen@gmail.com
2. Estudiantes, Departamento de Biología, Facultad de Ciencias Exactas y Naturales, Universidad del Valle, Cali, Colombia. e-mail: jco_933@yahoo.com angelitap@hotmail.com hegarces@hotmail.com
3.Profesor Titular, Sección de Genética, Departamento de Biología, Facultad de Ciencias Exactas y Naturales, Universidad del Valle, Cali, Colombia. e-mail: barretog@univalle.edu.co
Recibido para publicación julio 27, 2007 Aceptado para publicación abril 18, 2008
RESUMEN
Introducción: Estudios preliminares han mostrado la existencia de relaciones genéticas entre las poblaciones humanas del sur-occidente y las de la región andina colombiana, teniendo esto implicaciones en el grado de miscegenación de estas comunidades. No obstante el reconocimiento de este amplio proceso de mestizaje, no se tiene suficiente información que permita establecer la estructura y el grado de diversidad genética para cada región en particular y de la población colombiana en general.
Objetivo: Determinar la estructura y diversidad genética presente en grupos poblacionales del centro y sur-occidente colombiano.
Metodología: Se analizaron las frecuencias alélicas de 12 sistemas de microsatélites autosómicos y el tipo y frecuencia de RFLP’s de mtDNA presentes en 472 individuos de tres grupos étnicos: mestizos, indígenas y afroamericanos.
Resultados: La caracterización de haplotipos de mtDNA en individuos afrodescendientes presentó 15% de marcadores típicos amerindios y 43% de africanos. El análisis de la diversidad genética mostró un índice de 0.72 en individuos Pijaos, valor cercano al índice de diversidad de la población mestiza de Cali (0.75). El análisis molecular de varianza (AMOVA) a partir de los 12 STR’s, mostró que la estructuración genética no es significativa (FST de 0.032); adicionalmente se evidenció alta endogamia en la muestra mestiza de Caldas (0.43) y en la muestra indígena Coyaima (0.34).
Conclusiones: Con los marcadores moleculares estudiados se estableció la estructura genética de poblaciones del sur-occidente colombiano confirmándose adicionalmente el grado de miscegenación y el flujo genético ocurrido entre diferentes grupos étnicos del centro y sur-occidente colombiano.
Palabras clave: mtDNA; STR’s; Poblaciones amerindias; Diversidad genética; Miscegenación; Flujo génico; Sur-occidente colombiano.
Genetic diversity in human populations of two regions from Colombia
SUMMARY
Introduction: Preliminary studies have showed close relations among southwest human populations and Andean region leaving it consequences in ethnic admixe process. However, this wide process of racial admixture today it is not exist sufficient information to define structure and genetic diversity for each region and Colombian population in general.
Objectives: The principal goal from this study was to determinate the genetic structure and diversity present into human populations from Andean and Southwest Colombia regions.
Methods: This study was realized by characterization allelic frequencies of 12 autosomal STR’s and six RFLP’s of mtDNA presents in 472 individuals from three ethnics groups: Caucasoids, Afroamericans and Amerindians.
Results: mtDNA haplotypes presents in Afrodescends sample was 15% and 43% typical Amerindian and African markers respectively; the genetics diversity analysis shows a value of 0.72 in Pijao indigenous, these values are close to diversity index of mestizos from Cali (0.75). AMOVA of allelic frequencies from 12 STR’s shows that genetic structures don’t was significatively different (FST de 0.032); in addition it’s to exhibit high endogamy in mestizos from Caldas sample (0.43) and Coyaima indigenous (0.34).
Conclusions: was established genetic structure for southwest Colombian population. Additionally, the results confirm the mixing process and the genetics flow among many populations groups from Andean and southwest Colombia regions.
Keywords: mtDNA; STR’s; Amerindian populations; Genetics diversity; Mixed population; Genetics flow; Colombia southwest.
Las poblaciones latinoamericanas, se caracterizan por ser híbridas o mezcladas, con aportes genéticos de individuos pertenecientes a diversos linajes que colonizaron este vasto territorio utilizando varias rutas geográficas. Como resultado de estos eventos migratorios y seguro de otras interacciones socio-culturales, existe hoy una gran entremezcla racial compuesta sobre todo por amerindios, afrodescendientes y europeos, llegados de España y Portugal principalmente.
En Colombia dos de las regiones geográficas en donde se observa a nivel fenotípico, una aparente variación de la estructura genética de sus componentes poblacionales son el sur-occidente y el centro del país, porque estas zonas en épocas prehispánicas estuvieron ocupadas por diversas comunidades amerindias que fueron sometidas sistemáticamente hasta la desaparición casi total de las mismas por los europeos durante la colonia en los siglos XV y XVI.
Se justifica el uso de enfoques genético-moleculares para el estudio de la estructura genética e historia evolutiva en estas comunidades humanas por la presencia de diferentes grupos étnicos en una misma región geográfica o en regiones aledañas. En este sentido el empleo de marcadores de tipo biparental como los microsatélites o secuencias cortas repetidas en tandem (STR´s)1 y la caracterización de marcadores uniparentales generados a partir de los polimorfismos de tamaño de fragmentos de restricción (RFLP´s) en la molécula de ADN mitocondrial (ADNmt)2, constituyen herramientas válidas para caracterizar tanto el tipo como la frecuencia de alelos y haplotipos, así como para identificar la diversidad y el grado de estructura genética presente en las poblaciones consideradas en esta clase de estudios.
En Colombia, el conocimiento de la estructura genética de las poblaciones del centro y sur-occidente sigue siendo escaso a pesar de que se han realizado trabajos encaminados a establecer las frecuencias alélicas y haplotípicas de RFLP´s de ADNmt3-6 (en comunidades indígenas3-5 y afrodescendientes6), de STR´s autosómicos8, incluido el cromosoma Y4, 7, y evaluado el poder de discriminación, heterocigosidad y desequilibrio de ligamiento (LD) de diversos STR’s autosómicos procedentes de aislados poblacionales de diferentes grupos étnicos8, 9.
En el presente trabajo se evaluó la estructura y diversidad genética a partir de la caracterización de los haplotipos de mtDNA y de los revelados por 12 sistemas de marcadores STR’s, en cinco comunidades amerindias, en cuatro aislados afrodescendientes y cuatro poblaciones mestizos-caucasoides pertenecientes al centro y sur-occidente colombiano.
MATERIALES Y MÉTODOS
Poblaciones estudiadas. En total se estudiaron 477 individuos no relacionados, pertenecientes a tres grupos étnicos de diferentes departamentos del centro y sur-occidente colombiano. La muestra amerindia estuvo representada por 50 individuos Coyaimas y 35 Pijaos (Tolima); 42 Awa-Kuaikier (Nariño); 21 Paeces y 23 Yanaconas (Cauca) y 18 Emberás-Dumá (Chocó). La muestra mestiza estuvo representada por 27 personas del departamento de Cauca, 21 de los departamentos del eje cafetero, 103 de Santiago de Cali y 69 de otros muni-cipios del Valle del Cauca. La muestra afrodescendiente estuvo representada por 5 individuos de Bahía Solano (Chocó); 4 de Tumaco (Nariño); 19 de Güapi (Cauca) y 40 de Buenaventura (Valle). Las muestras mestizas y afrodescendiente fueron obtenidas del Servicio de Extensión del Laboratorio de Genética Molecular Humana de la Universidad del Valle, mientras que las indígenas se escogieron por el aislamiento geográfico de las mismas. En todos los casos los participantes en este estudio firmaron un informe de consentimiento antes de tomar los 5 ml de sangre periférica. A partir de esta muestra se extrajo el ADN genómico por el método de desalamiento (Salting out)10 con el objetivo de caracterizar los diferentes marcadores moleculares y a partir de ellos evaluar la diversidad genética presente en los aislados poblacionales considerados.
Polimorfismos de mtDNA. Con el objetivo de identificar la ancestría de las muestras, se buscaron los cuatro linajes de haplotipos fundadores de poblaciones amerindias (A-D)2 y el haplotipo denominado «E»11 en las muestras indígenas Pijao y Emberá-Dumá, así como en 32 mestizos-caucaosides del municipio de Versalles y 43 afrodescendientes del aislado poblacional de Mulaló (Valle). Las muestras de individuos Awa, Coyaima, mestizos de Cali y la totalidad de los individuos afrodescendientes ya habían sido caracterizados para los RFLP’s de mtDNA9; además en aquellos individuos que no presentaron los marcadores típicos de amerindios, se caracterizaron marcadores típicos de haplotipos africanos12. Los iniciadores, las condiciones de amplificación por PCR (Minicycler, MJ Research) y las condiciones de restricción enzimática se realizaron como está descrito en la literatura9, 11.
Marcadores microsatélites autosómicos. Con el objetivo de evaluar el aporte biparental presente en las comunidades estudiadas, se estimó las frecuencias alélicas de 12 sistemas STR’s autosómicos de uso convencional en estudios de filiación y genética forense. Se analizaron los siguientes STR´s HUMTH0113, FES13, D7S82014, D13S31714, HumvWA15, HumFGA15, F13A0116, CSF1PO17, D21S1118, F13B19, LPL20 y TPOX21 los cuales se amplificaron por la PCR utilizando las condiciones de reacción descritas en la literatura para cada sistema.
Análisis de los resultados. Los productos amplificados de los haplotipos mitocondriales, los RFLP’s y los sistemas STR’s nucleares se separaron en geles de poliacrilamida al 8%, teñidos con nitrato de plata. La asignación alélica para los microsatélites se realizó utilizando escaleras alélicas para cada sistema, confirmándose luego estos resultados mediante la utilización del sistema automatizado de análisis UVISave (UVITec Limited, BL WA, Inglaterra).
Análisis de los datos. La diversidad genética se estimó como [n/(n-1)] (1-Ópi2), donde n es el tamaño de muestra y pi es la frecuencia alélica o haplotípica esti-mada del alelo o haplotipo i22. Se empleó el software ARLEQUÍN Versión 3.123 para calcular la distancia genética a partir de parejas de FST, con significancia diferente de cero evaluada por una prueba de aleatorización.
RESULTADOS
Haplotipos de mtDNA. La muestra poblacional de Versalles presentó para el haplotipo B una frecuencia de (80%) mientras que 94% de la Pijao se distribuyó entre los haplotipos A, B y C. Se destacó también en esta última comunidad el haplotipo G (origen asiático) con una frecuencia de 11%. La muestra afrodescendiente de Mulaló, presentó para el haplotipo L (origen africano) una frecuencia de 67% mientras que en 15% del total de la muestra afrodescendiente se observaron los haplotipos A, B y C típicos de amerindios.
Las frecuencias de los haplotipos mitocondriales, característicos de las comunidades indígenas, identificados en las poblaciones Awa, Coyaima, Emberá-Dumá y mestizos de Cali habían sido establecidas previamente por nuestro grupo9, siendo utilizadas en este trabajo para determinar distancias, diversidad y estructura genética de las poblaciones en estudio.
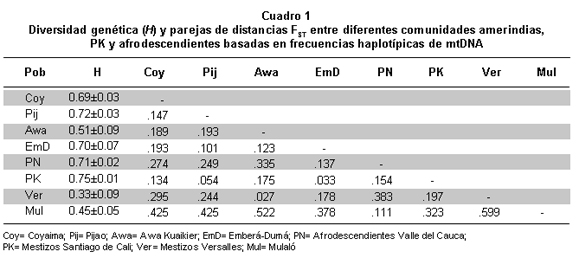
Distancias y diversidad genética de haplotipos de mtDNA. El Cuadro 1 registra la diversidad y las distancias genéticas basadas en las frecuencias de los haplotipos de mtDNA en las poblaciones analizadas en el presente estudio. En cuanto a las distancias genéticas, se destaca el hecho de que todas las muestras de las comunidades indígenas estudiadas presentaron las mayores distancias con respecto a la muestra de Mulaló; también se destaca la relación de cercanía de la muestra de mestizos de Cali con los aislados Emberá-Dumá (.033) y Pijao (.054), mientras que la muestra de mestizos de Versalles es más cercana a Awa Kuaikier (.027) que a la otra muestra mestiza considerada (.197). Este hecho evidencia la separación de estas dos muestras poblacionales que se encuentran ubicadas dentro de la misma región geográfica, como sucedió también con la muestra indígena Coyaima, que está más cercana a la muestra mestiza de Cali (.134) que a la muestra Pijao (.147). Como era de esperarse, las dos muestras afrodescendientes exhibieron la menor distancia genética (.111) entre ellas.
Los resultados permitieron observar que la menor diversidad genética se presentó en la muestra de Versalles (0.33) mientras que la más alta se presentó en los mestizos de Cali (0.75), hecho contrastante para dos poblaciones mestizo-caucasoides. También es notoria la diferencia en los valores de diversidad genética presentes entre Mulaló (0.45) y el resto de la población afrodescendiente estudiada (0.71). Entre las comunidades indígenas, Awa presentó la menor diversidad genética (0.51) y Pijao la mayor (0.72). Este último valor es relativamente cercano a la diversidad genética de la población mestiza de Cali (0.75) y a la de la población mezclada de Santafé de Bogotá (0.76)6 y se encuentra dentro del rango de valores registrado para los Ticuna9 (0.71) y los Murui-Muinane y Piaroa5 (0.78).
Diversidad y estructura genética de microsatélites autosómicos. En el Cuadro 2 se presentan los valores de heterocigosidad promedio de cada uno de los aislados poblacionales a partir de las frecuencias alélicas de 12 sistemas de microsatélites autosómicos. De estos valores se deduce que la mayor y menor diversidad genética se observó en las muestras afrodescendientes del Cauca (0.93) y de Nariño (0.68) respectivamente; también se visualizó que la diversidad genética de las muestras mestizas se distribuyeron entre 0.81 (Palmira) y 0.91 (Cauca). Los valores de diversidad en las muestras mestizas y afrodescendientes del departamento del Cauca, podrían estar en relación con la alta diversidad observada en las etnias indígenas Paez (0.87) y Yanaconas (0.85); estos valores, a su vez, son los más altos comparados con las otras poblaciones amerindias analizadas (Cuadro 2). Estos resultados en general, aportan evidencia adicional al entendimiento del proceso de mestizaje que han venido sufriendo las comunidades indígenas mencionadas, aspecto que se había empezado a observar en Coyaima y en Pijao cuando se analizaron diez sistemas STR’s autosómicos9.
Con el fin de conocer si existen diferencias significativas en las frecuencias alélicas de los grupos poblacionales estudiados, se realizó un análisis jerarquizado de los datos, donde se cotejaron las varianzas para cada uno de ellos. Para esto se compararon las 22 poblaciones en un grupo general y en los tres grupos étnicos y con estos datos se realizó el análisis molecular de varianza (AMOVA) que muestra el índice FST (un grupo), y los índices FSC y FCT para los grupos étnicos evaluados.
El valor de diferenciación subpoblacional o FST fue de 0.32 cuando se realizó el análisis de un solo grupo; este valor queda representado en los valores FSC y FCT producto de la comparación de los tres grupos étnicos tal como lo muestra la AMOVA (Cuadro 3). Este análisis confirmó la existencia de diferencias significativas entre las poblaciones estudiadas, observándose un porcentaje de variación dentro de grupos equivalente a 2.8% debido a la diferencia entre poblaciones (FSC=0.028) correspondiente a las frecuencias alélicas observadas para los grupos étnicos. Se logró establecer que 96.6% de la variación es aportada por los individuos de cada población estudiada, confirmando el carácter polimórfico de los sistemas STR’s estudiados, corroborado en los valores de heterocigosidad o diversidad genética promedio (Cuadro 2). El porcentaje de variación para la estructura genética fue de 0.6%. Este valor evidencia el hecho de que los grupos étnicos comparados para el sur-occidente y centro colombiano no son diferentes entre sí en cuanto a su estructura genética.
El análisis de la endogamia presente en los 22 aislados poblacionales mostró valores positivos inferiores a 43%, excepto en los aislados de Restrepo y Bolívar (Valle) lo que indica apareamientos preferenciales entre individuos heterocigotos en estas poblaciones (Cuadro 2). Estos resultados podrían estar siendo influidos por los bajos tamaños de la muestra en algunas poblacionales.
Basado en las parejas de distancias genéticas FST de los 12 sistemas estudiados para cada uno de los grupos poblacionales muestriados, se realizó un análisis de inferencia filogenética por el método de los vecinos cercanos o Neighbor-Joining (NJ) en el software Mega 3.124 (Figura 1). El árbol mostró una relación, de manera acentuada, entre las muestras afrodescendientes de Nariño, Cauca y Valle con las muestras amerindias Paeces y Yanaconas. La población mestiza de Cali y del resto del Valle también presentó fuerte relación con el resto de las muestras indígenas analizadas, confirmando la observación realizada a partir de las distancias genéticas de los marcadores de mtDNA, en donde se establece la cercanía de estas comunidades con las muestras mestizas. Llama la atención el hecho de que las poblaciones del centro y norte del Valle se separan de Cali y el resto del Valle, en una misma agrupación junto con las muestras de Popayán y mestizos de Cauca, Risaralda y Caldas; este resultado es evidencia de la conectividad poblacional que existe entre comunidades del centro y del sur-occidente colombiano.
DISCUSIÓN
El 80% de frecuencia del haplotipo B encontrado en la población de Versalles es el valor más alto hallado para este marcador mitocondrial en muestras poblacionales colombianas no indígenas5, 6, 9; por otro lado, la frecuencia de 67% para la muestra afroamericana de Mulaló también es la más alta comparada con la misma etnia en la isla de Providencia (52.4%)6, San Basilio de Palenque (Bolívar) (44.7%)6 y Pacífico colombiano (29%, presente estudio), resultados que posiblemente estarían reflejando niveles de endogamia importantes al interior de estas dos poblaciones6.
Los polimorfismos de mtDNA caracterizados en las muestras mestizas de Versalles continúan apoyando la hipótesis del origen matrilíneo amerindio en esta población. Esta hipótesis surgió cuando se analizó la presencia de marcadores de este tipo en la población mezclada del departamento de Antioquia4 y se corrobora con resultados similares en el análisis de la población mezclada de Bogotá5 y Cali9. No obstante estos resultados, no se puede descartar la idea de la presencia de haplotipos mitocondriales típicos de poblaciones europeas, dada su cercanía con los departamentos de Caldas, Risaralda y Quindío de donde se conoce partieron, aproximadamente hace 90 años, los fundadores de la actual comunidad de Versalles. Esto traería como consecuencia el aumento, en cierto grado, de la diversidad genética en esta comunidad por el bajo valor de heterocigosidad promedio exhibido.
La presencia del haplogrupo C podría ser relevante al momento de evaluar las distancias genéticas, como sucede al comparar los mestizos de Cali con Awa9; este efecto no se logra observar en Versalles porque en esta muestra sólo se caracterizaron los haplotipos A (20%) y B (80%), que también fueron caracterizados en todas las muestras amerindias consideradas. Otro hecho que se debe considerar, es la ausencia del haplotipo E en las comunidades Pijao y Emberá-Dumá, lo que no sucede en Awa y Coyaima9, que podría estar generando un claro distanciamiento entre éstas últimas con respecto a las demás comunidades amerindias consideradas, además de generar evidencia de un evento reciente de aparición de este marcador en comunidades amerindias como fue planteado en un estudio realizado en grupos poblacionales del desierto de Atacama11.
En un trabajo anterior se había planteado que tanto las poblaciones mestizas como las afrodescendientes que se analizaron, presentaron mezcla asimétrica6. Esta misma apreciación se logra observar con los resultados obtenidos en muestras afrodescendientes y mestizas del Valle del Cauca9 y se ratifica con la presencia del haplotipo africano L en 67% de la muestra de Mulaló, porque permite suponer la presencia de marcadores típicos Amerindios en el resto de la muestra de este aislado poblacional, lo cual estaría reflejando el establecimiento de apareamientos entre varones de origen africano con mujeres indígenas establecidas en dicha zona en épocas del desarrollo de la esclavitud, en los siglos XVII a XIX en el occidente colombiano25.
En cuanto a los sistemas STR’s analizados, ya se había advertido la caracterización de valores altos de las frecuencias alélicas de algunos sistemas9, particularmente TH01, que mostraría un proceso asociado con homocigosis para las muestras poblacionales que las exhiben. Esta observación estaría directamente relacionada con el alto índice de endogamia (Cuadro 2) presente en comunidades indígenas como Awa Kuaikier y Coyaima, corroborando el hecho de que las poblaciones indígenas muestran alelos representativos9. Este hecho se puede sustentar en el registro histórico de las poblaciones indígenas, en donde las prácticas matrimoniales incluyen el establecimiento de castas como sucede en Awa Kuaikier en donde cada tres generaciones hay presencia de endogamia intensiva26. Esta observación refleja la poca relación con las muestras poblacionales mestizas o negras consideradas en el presente estudio, sin ser totalmente concluyente y concomitante para otras poblaciones indígenas como los Yanaconas y los Paeces (Figura 1).
Las muestras poblacionales mestizas aquí estudiadas mostraron índices de diversidad génica superiores a 80% para 12 sistemas STR’s, comparado con el índice de diversidad en población mezclada en el departamento de Antioquia (0.79) a partir del análisis de 20 STR’s autosómicos7. Los resultados de diversidad tanto en las poblaciones mestizas como afrodescendientes analizadas para el sur-occidente y el centro de Colombia, podrían deberse principalmente a la presencia de variantes alélicas diferentes para alguno de los sistemas considerados, así como a los pequeños tamaños de muestra analizada. Sin embargo, la escasa diferenciación genética (FST=0.032) se debe a que se ha presentado un constante flujo genético entre poblaciones del centro y del sur-occidente colombiano. Este flujo genético estaría involucrando los tres grupos étnicos principales presentes en estas regiones de Colombia. Un sustento adicional a este planteamiento, se logra evidenciar en la conexión, pese a la separación en dos grupos filéticos, de ocho poblaciones del sur-occidente y del centro del país estudiadas para seis sistemas de microsatélites (Figura 2).
CONCLUSIONES
Las poblaciones humanas analizadas pese a exhibir alta diversidad genética mitocondrial y nuclear, se comportan como una misma unidad en cuanto a su estructura genética, por el bajo valor FST encontrado con el análisis molecular de varianza de las frecuencias alélicas de los 12 sistemas STR’s analizados; estas observaciones son evidencia de la conectividad presente entre las poblaciones del centro y sur-occidente colombiano. Además, se demuestra la mezcla asimétrica que los tres grupos étnicos principales aportan a dichas comunidades en la cual se refleja que entre 80% y 15% de las muestras mestizas y afrodescendientes, respectivamente, tienen origen amerindio y por ende asiático, mientras que el resto del aporte es mayoritariamente africano y en segunda instancia europeo, con lo cual se pueden realizar asignaciones más precisas al analizar la diversidad de condiciones genéticas heredables implicadas en enfermedades y sobre incidencia los diferentes componentes poblacionales de una región geográfica en particular.
AGRADECIMIENTOS
El Grupo de Investigación en Genética Molecular Humana, Universidad del Valle, Cali, quiere reconocer el aporte hecho por las personas que donaron las muestras de sangre para el éxito de este estudio. También agradecemos al señor Orlando Sánchez, Presidente de Afro-Yumbo, por autorizar la realización de este estudio en la comunidad de Mulaló y a la profesora Esmeralda Ortiz por la colaboración en el acercamiento a esta comunidad; a la profesora Blanca Leonor López por permitir la obtención de las muestras de la comunidad de Versalles. A los doctores Héiber Cárdenas y Esteban Osorio, profesores de la Sección de Genética, Universidad del Valle, por las asesorías en el análisis de los resultados y el préstamo de equipos. A la Vicerrectoría de Investigaciones de la Universidad del Valle por la financiación parcial del proyecto de investigación del cual hace parte el presente estudio y a COLCIENCIAS por el otorgamiento de la beca doctoral a Fernando Rondón.
REFERENCIAS
1. Ruiz-Linares A. Microsatellites and the reconstruction of the history of human populations. En: Goldstein DB, Schlotterer C, editores. Microsatellites: evolution and applications. Oxford: Oxford University Press; 1999.
2. Torroni A, Schurr TG, Cabell MF, Brown D, Neel JV, Larsen M, et al. Asian affinities and continental radiation of the four founding native american mtDNAs. Am J Hum Genet. 1993; 53: 591-608.
3. Rondón F, Vallejo G, Osorio E, Barreto G. Frecuencias haplotípicas mitocondriales en la comunidad indígena Coyaima del departamento del Tolima, Colombia. Rev Asoc Colomb Cienc Biol. 1999; 11: 45-53.
4. Mesa NR, Mondragón MC, Soto ID, Parra MV, Duque C, Ortiz-Barrientos D, et al. Autosomal, mtDNA and Y-chromosome diversity in Amerinds: Pre- and post-Columbian patterns of gene flow in South America. Am J Hum Genet. 2000; 67: 1277-86.
5. Keyeux G, Rodas C, Gelvez N, Carter D. Posible migration routes into South America deduced from mitochondrial DNA studies in Colombian Amerindian populations. Hum Biol. 2002; 74: 211-33.
6. Rodas C, Gelvez N, Keyeux G. Mitochondrial DNA studies show asymmetrical Amerindian admixture in Afro-Colombian and Mestizo populations. Hum Biol. 2003; 75: 13-30.
7. Carvajal-Carmona LG, Soto ID, Pineda N, Hartiala J, Molina J, León P, et al. Strong Amerind/White sex bias and a posible Sephardic contribution among the founders of a Population in Norwest Colombia. Am J Hum Genet. 2000; 67: 1287-95.
8. Gómez MV, Reyes ME, Mejía DF, Cárdenas H. Genetic variation at 9 STR loci in a southwestern Colombian population. Prog Forens Genet. 2000; 8: 251-3.
9. Rondón F, Orobio RF, Braga Y, Cárdenas H, Barreto G. Estudio de la diversidad genética de cuatro poblaciones aisladas del centro y sur-occidente colombiano. Rev Salud UIS. 2006; 38: 9-15.
10. Gustincich S, Carminci P, Del Sal G, Mamfiolelle G, Schneider C. A fast method for high-quality genomic DNA extraction from whole human blood. Biotechniques. 1989; 11: 25.
11. Bailliet GF, Rothhammer F, Carnese FR, Bravi CM, Bianchi NO. Founder mitochondrial haplotypes in American populations. Am J Hum Genet. 1994; 54: 27-33.
12. Denaro M, Blanc H, Johnson MJ, Chen KH, Wilmsen E, Cavalli-Sforza LL, et al. Ethnic variation in HpaI endonuclease cleavage patterns of human mitochondrial DNA. Proc Nat Acad Sci USA. 1981. 78: 5768-72.
13. Polymeropoulos MH, Rath S, Xiao H, Merril CR. Tetranucleotide repeat polymorphism at the human coagulation factor XIII a subunit gene (F13A01). Nucleic Acid Res. 1991; 19: 4306.
14. Jin L, Underhill PA, Buoncristiani MR, Robertson JM. Defining microsatellite alleles by genotyping global indigenous human populations and non-human primates. J Forensic Sci. 1997; 42: 496-99.
15. Urquhart A, Oldroyd NJ, Kimpton CP, Gill P. Highly discriminating heptaplex short tandem repeat PCR system for forensic identification. Biotechniques. 1995; 18: 116-28.
16. Edwards A, Civitello A, Hammond HA, Caskey CT. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats. Am J Hum Genet. 1991; 49: 746-56.
17. Ruitberg CC, Reeder DJ, Butler JM. STRBase: a short tandem repeat DNA database for the human identity testing community. Nucleic Acid Res. 2001; 29: 320-2.
18. Sharma V, Litt A. Tetranucleotide repeat polymorphism at the D21S11 locus. Hum Mol Genet. 1992; 1: 67.
19. Nishimura DY, Murray JC. A tetranucleotide repeat for the F13B locus. Nucleic Acids Res. 1992; 20: 1167.
20. Zuliani G, Hobbs HH. Tetranucleotide repeat polymorphism in the LPL gene. Nucleic Acid Res. 1990; 18: 4958.
21. Anker R, Steinbrueck T, Donis-Keller H. Tetranucleotide repeat polymorphism at the human thyroid peroxidase. Hum Mol Genet. 1992; 1: 137.
22. Nei M. Molecular evolutionary genetics. New York: Columbia University Press; 1987.
23. Excoffier L, Laval G, Schneider S. ARLEQUIN an integrated software package for population genetics data analysis [programa de computador]. Versión 3.1. Berne: University of Berne; 2006.
24. Kumar S, Tamura K, Nei M. MEGA3: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinfor. 2004; 5: 150-63.
25. Zapata, M. La más vieja raíz de la humanidad. En: La rebelión de los genes. El mestizaje americano en la sociedad futura; Bogotá, DC: Altamir ediciones; 1997. p. 71-144.
26. Cerón B. Elementos que permiten acercarnos a la evolución histórica del grupo indígena Kwaiker. En: ABYA-YALA (eds). Los Awa-Kuaiker, un grupo indígena de la selva pluvial del Pacífico nariñense y el nor-occidente ecuatoriano. Quito: Editorial ABYA-YALA; 1988. p. 195-238.