Tratamiento con imatinib y el farmacogenotipo CYP3A4 en relación con la expansión clonal Ph(+) en leucemia mieloide crónica (LMC)*
Mauricio Camargo, PhD1, María Isabel Soto-Marín, Biol1, Olga Zea, Biol1, Domingo Saavedra, MD2
* Este artículo contó con el apoyo económico del Comité para el Desarrollo de la Investigación (CODI), Acta N° 66, 31-05-06, Universidad de Antioquia, Medellín, Colombia.
1. Grupo de Genética de Poblaciones y Mutacarcinogénesis, Sede de Investigación Universitaria (SIU), Universidad de Antioquia, Medellín, Colombia. e-mail: mcamargo@une.net.co maria_soto_m@yahoo.com
olz.geneticalab@une.net.co saavedra-rueda@une.net.co
2. Médico hematólogo, Clínica Vida, Medellín, Colombia.
Recibido para publicación marzo 11, 2008 Aceptado para publicación septiembre 18, 2008
RESUMEN
Introducción: imatinib es un inhibidor de la tirosina-kinasa BCR-ABL que revolucionó el tratamiento de pacientes con leucemia mieloide crónica (LMC) positivos para cromosoma Philadelphia (Ph+). Este medicamento se metaboliza principalmente por la enzima CYP3A4, cuyo gen presenta variaciones interindividuales tipo SNPs que pueden interferir con la efectividad del tratamiento, como son los polimorfismos CYP3A4*1B y CYP3A4*2 que han mostrado influencia significativa en la actividad metabólica de esta importante enzima farmacológica.
Objetivos: Evaluar la frecuencia de polimorfismos de importancia farmacogenética en el gen CYP3A4 en una población de pacientes con LMC tratados con imatinib y en una población control de 164 personas. Correlacionar el genotipo con la evolución de la expansión clonal Ph(+) y la duración del tratamiento.
Metodología: Genotipificación PCR-RFLP para los SNPs CYP3A4*1B y CYP3A4*2. Bandeo replicativo tipo RBHG para la evaluación citogenética de blastos espontáneos con o sin presencia del marcador Ph(+).
Resultados: Los análisis citogenéticos revelaron una correlación directa entre el tiempo de tratamiento con imatinib y el porcentaje de reducción de blastos Philadelphia (+). Las genotipificaciones evidenciaron que la presencia del polimorfismo CYP3A4*1B no influye en la respuesta citogenética de los pacientes Ph+ tratados con imatinib, y que el polimorfismo fármaco relevante CYP3A4*2 está ausente en esta población colombiana de controles y pacientes.
Conclusiones: El farmacogenotipo CYP3A4*2 (exón 7) no afecta la respuesta citogenética positiva inducida por el imatinib en pacientes con LMC, en quienes la frecuencia de células Ph(+) por lo general se reduce en relación directa con la duración del tratamiento.
Palabras clave: Imatinib; Leucemia mieloide crónica; CYP3A4; Cromosoma Philadelphia; Farmacogenética; SNPs.
Imatinib treatment and pharmacogenotype CYP3A4 in relation with the clonal expansion Ph(+) in chronic myeloid leukemia (CML)
SUMMARY
Introduction: Imatinib is an inhibitor of the BCR-ABL tyrosine-kinase that has dramatically changed the treatment of patient with Chronic myeloid leukemia (CML) positive for the Philadelphia chromosome (Ph+). This compound is mainly metabolized by the cytochrome CYP3A4 enzyme, coded by a gene with individual variations that could interfere with the effectiveness of the treatment, due to the fact that particular single nucleotide polymorphisms (SNPs), i.e., CYP3A4*1B y CYP3A4*2, have shown to exert a significant influence on the metabolic activity of this pharmacologically important enzyme.
Objective: Evaluate the frequency of pharmacogenetically important polymorphisms in the CYP3A4 gen in a Colombian population of patients with CML being treated with this novel drug (Imatinib), in parallel with a control population of 164 healthy individuals. Correlate the evolution of the clonal expansion Ph(+) with the presence of these SNPs and the length of treatment.
Methodology: PCR-RFLP genotyping for the CYP3A4* 1B y CYP3A4*2 SNPs. RBHG replication banding for the evaluation of the presence of the Ph(+) markers in spontaneous mitotic blasts.
Results: A positive cytogenetic response and/or correlation was detected between the length of the imatinib treatment and a reduction in the percentage of Ph(+) blasts. Genotyping indicate that CYP3A4*1B polymorphism does no affect the cytogenetic response in imatinib treated Ph(+) patients, and that the pharmacorelevant CYP3A4*2 SNP is not present in this population of patients and controls (N=194).
Conclusions: The pharmacogenotype CYP3A4*2 (exon 7) does not affect the induced positive cytogenetic response triggered by the imatinib treatment, that generally induces a reduction in Ph(+) blasts en relation with the duration of the treatment.
Keywords: Imatinib; Chronic myeloid leucemia; CML; CYP3A4; Philadelphia chromosome; Pharmacogenetics; SNPs.
La leucemia mieloide crónica (LMC) es un desorden mieloproliferativo clonal en células madre hematopoyéticas, estrechamente ligado con la aparición del cromosoma Philadelphia que resulta de una translocación recíproca entre los cromosomas 9 y 22 t(9;22) (q34;q11), y según la región donde ocurra el rearreglo genómico, puede dar origen a varios tipos de proteínas de fusión BCR-ABL, siendo las más comunes la 210, 190 y 230 kDa. La actividad tirosina-kinasa de estas proteínas se asocia con una señalización compleja que se traduce en un fenotipo celular alterado o transformación maligna por interferencia con procesos celulares básicos que incluyen señalización mitogénica constitutivamente activa, adherencia defectuosa a células estromales y matriz extracelular, además de una reducida respuesta a estímulos inductores de apoptosis. Por tal motivo, a esta translocación se le adjudica un importante papel en la regulación y diferenciación ciclo-dependiente1-2.
El curso de la enfermedad es característicamente trifásico: la mayoría de los pacientes pasan por una fase crónica prolongada, seguida de una transformación o aceleración de la enfermedad con progresiva resistencia al tratamiento y al final una fase blástica de muy corta duración2. Es una leucemia potencialmente curable con transplante alogénico de células madre, pero menos de 30% de los enfermos tienen donantes compatibles. De otro lado, la quimioterapia con interferón-alfa sólo produce una respuesta citogenética completa entre 5% y 20% de los pacientes, y por lo general, está asociada con serios efectos tóxicos. La terapia alternativa con hidroxiurea o busulfan, sólo alcanza una respuesta hematológica cercana a 50%. Además, la tasa de respuesta disminuye rápido cuando estos segundos tratamientos se inician en la última etapa de la fase crónica de la enfermedad (aproximadamente 12 meses después del diagnóstico inicial).
En contraste, el imatinib ha revolucionado la quimioterapia de la LMC. Es uno de los primeros medicamentos antineoplásicos desarrollados para un blanco molecular específico inhibidor de transducción de señales. Su principio activo es el mesilato de imatinib, que actúa bloqueando el sitio de unión a ATP en la proteína kinasa ABL, inhibiendo su capacidad para transferir un grupo fosfato desde el ATP y fosforilar residuos de tirosina en proteínas sustrato, lo que impide la transducción de señales necesarias para inducir proliferación celular y apoptosis. Su actividad terapéutica se ha extendido y con éxito a otros trastornos neoplásicos como el tumor estromal gastrointestinal kit (CD117+)3, síndrome de hipereosinofilia idiopática4, leucemia linfoide aguda Ph(+)5, y mastocitosis sistemica6 pues el imatinib resultó ser un inhibidor selectivo de otras tirosina-kinasas como PDGF-R y C-kit involucradas en estas enfermedades7,8.
El mesilato de imatinib es metabolizado principalmente por la enzima CYP3A4 del sistema enzimático citocromo P450 (CYP) cuyo gen está localizado en el locus cromosómico 7q21.1 (OMIM124010) y es clasificado como polipéptido 4 de la subfamilia IIIA, superfamilia citocromo P4509. Su principal metabolito es la N-desmethylated piperazine que posee una actividad in vitro comparable con la del imatinib. Pero un factor importante que afecta el buen desarrollo de la farmacoterapia CYP-dependiente, es el descubrimiento de variaciones o polimorfismos en dichos genes, que como los de la familia del citocromo P450, están involucrados en el metabolismo de la mayoría de la quimioterapia humana actual10.
Entre los polimorfismos farmaco-relevantes está el SNP CYP3A4-V, también llamado CYP3A4*1B, que consiste en una sustitución A ----> G en la posición -290 de la región promotora del gen11, que ha revelado diferencias funcionales con su contraparte «silvestre»12. Otro polimorfismo importante es el CYP3A4*2 que consiste en una sustitución T673C en el exon 7 del gen, que genera un sitio de reconocimiento para la endonucleasa MnlI13. Este polimorfismos (Ser®Pro) ha mostrado diferencias cuantitativas en la actividad enzimática14 debido a que el cambio de serina por prolina suele alterar la estructura tridimensional de la enzima, porque la prolina suele generar un giro o codo en la hélice de la proteína. En consecuencia, y por la documentada ingerencia de los farmacogenotipo sobre la quimioterapia individualizada, el principal objetivo del presente estudio fue evaluar la influencia que puedan tener importantes SNPs del farmacogen CYP3A4, sobre la evolución clonal Ph(+) y duración del tratamiento en personas con LMC.
MATERIALES Y MÉTODOS
Muestras de ADN. Durante un año se colectaron muestras de ADN a partir de 3-5 ml de sangre periférica de un total de 30 personas con diagnóstico de LMC y 164 voluntarios sanos quienes previamente leyeron y firmaron el respectivo consentimiento informado, aprobado por el Comité de Ética de la Sede de Investigaciones Universitarias (SIU) de la Universidad de Antioquia, Medellín, Colombia. Las muestras se almacenaron refrigeradas hasta el momento del análisis. La extracción del ADN se hizo por el método salino15 y el ADN rehidratado y cuantificado se almacenó a -20°C.
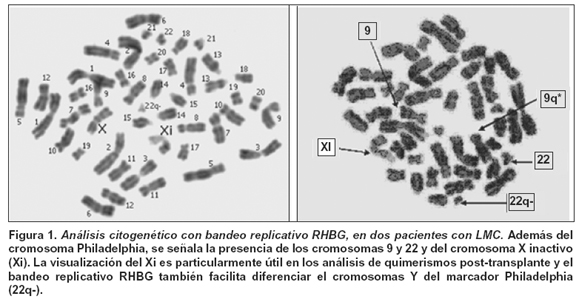
Cultivos celulares y análisis citogenético. Con el fin de determinar el porcentaje de células positivas para el cromosoma biomarcador Phliladelphia, a cada uno de los pacientes con diagnóstico de LMC se les realizó análisis citogenético con bandeo replicativo RHBG (bandeo R con Hoechst-BrdU-Giemsa) en linfocitos de sangre periferical. Se sembraron 4-6 gotas de sangre total en 9 ml de medio de cultivo F12 suplementado con SBF 10%, y en ausencia de mitógeno externo. El patrón replicativo RBHG se obtuvo mediante pulso terminal de 7 horas con BrdU (20 mg/ml) y colchicina (0.4 mg/ml), y la coloración diferencial respectiva (Figura 1)16.
Genotipificación. Se genotipificaron los dos polimorfismos más importantes de un solo nucleótido (SNP) que afectan la actividad enzimática de CYP3A4, principal metabolizador del Imatinib.
CYP3A4*1B (región promotora). La PCR para CYP3A4*1B se realizó en un volumen de 25 ml que contenía 50 ng de ADN geonómico, 2.5 l de buffer PCR 10X (500 mM KCl, 100 mM Tris-HCl, pH 9), 200 mM de dNTP, 0.8 mM de MgCl2, 1 U de Taq polimerasa y 0.4 µM de cada primer (5'-GGA CAGCCA TAG AGA CAA CTG CA-3') (5'-CTT TCC TGC CCT GCA CAG-3'). El perfil de amplificación fue 94°C (7') un ciclo, seguido por 35 ciclos a 94°C (1'), 54, 7°C (1'), 72°C (1') y una extensión final a 72°C (7'). Para esta región se obtiene un amplificado de 334 pb12. La digestión del amplificado (10 ml) se realizó en una reacción de 20 ml con 3 U de enzima PstI y 2 ml de buffer de digestión a 37°C 16 h.
CYP3A4*2 (Exón 7). La PCR para CYP3A 4*2 se realizó en un volumen de 25 ml que contenía 50 ng de ADN genómico, 1 mM de cada primer (5'-CCT GTT GCA TGC ATA TC-3'), (5'-GAT GAT GGT CAC ACA TAT C-3'), 2.5 ml de buffer PCR 10X (500 mM KCl, 100 mM Tris-HCl, pH 9,0), 200 mM de dNTP, 1.5 mM de MgCl2, y 1 U de Taq polimerasa. El perfil de amplificación fue 94°C (5') un ciclo, seguido por 35 ciclos a 94°C (1'), 60°C (1'), 72°C (1') y una extensión final a 72°C (10'). Para esta región se obtiene un amplificado de 366 pb. La digestión del amplificado (10 ml) se realizó en una reacción de 20 ml con 3 U de enzima MnlI y 2 ml de buffer de digestión, a 37°C 16 h. En ambos casos, los productos de la restricción se analizaron en un gel de agarosa al 3% (TBE 1X, 85 V, 40') con bromuro de etidio (Figura 2).
Análisis estadístico. La muestra de los pacientes fue de 30 individuos con diagnóstico confirmado de LMC y colectados en diferentes hospitales de la ciudad, canalizados a través del programa de subvención del imatnib por intermedio de Novartis-Pharma. La muestra control fue de 164 individuos sanos colectados con el propósito de estimar la frecuencia poblacional para los polimorfismos CYP3A4*1B y CYP3A4*2, y el tamaño se estimó de acuerdo con una frecuencia mínima esperada de p=0.05 (frecuencia del alelo más infrecuente), y q=1-p (para el alelo mayor) y bajo 95% de confiabilidad, y un porcentaje de error permitido de 5%. El porcentaje de blastos Ph(+) se determinó mediante análisis citogenético de cien blastos mitóticos bandeados con RBHG.
Para determinar si existe una correlación entre el porcentaje de permanencia del biomarcador Philadelphia y el tiempo de tratamiento con imatinib (meses), se realizó un análisis de regresión simple usando el software STATISTICA 6.0. De otro lado, para determinar si hay diferencias significativas en cuanto al porcentaje de Ph(+) con respecto a cada uno de los genotipos del polimorfismo CYP3A4*1B, se usó la prueba no paramétrica de Kruskal-Wallis (STATISTICA 6.0). Y para evaluar el efecto de la interacción tiempo-genotipo se usó un análisis ANOVA de dos factores.
RESULTADOS
Correlación tiempo de tratamiento vs. frecuencia del biomarcador cromosoma Philadelphia (Ph+). Los análisis citogenéticos para los pacientes con diagnóstico de LMC revelaron el valor porcentaje de blastos con cromosoma Philadelphia presente al momento del tratamiento cuando se realizó la toma de la muestra. Y el bandeo RBHG resultó de gran utilidad para identificar este marcador citogenético (Figura 1). Con el fin de establecer si existe o no una correlación entre el tiempo de tratamiento con imatinib y el porcentaje de blastos Ph(+), el análisis de regresión lineal mostró relación inversa (r=-0.5376) entre las dos variables analizadas; por tanto, se concluye que en la población analizada, a medida que aumenta el tiempo de tratamiento con imatinib, disminuye el porcentaje de células Ph(+) (Gráfica 1). Estos resultados concuerdan con estudios de respuesta citogenética a imatinib2 y son los resultados comúnmente esperados en una exitosa terapia farmacológica para la LMC. A continuación se evaluó la presencia de los farmacogenenotipos mencionados y su efecto sobre la terapia con imatinib.
Distribución de genotipos y frecuencias alélicas de polimorfismos CYP3A4 en las poblaciones de estudio. Como el estudio se realizó en pacientes de clínicas de Medellín, se utilizó una población control de la misma cuidad. La distribución de genotipos y alelos se muestra en el Cuadro 1. La frecuencia alélica encontrada para el polimorfismo CYP3A4*2 (Ser673 Pro) fue de 0.0 tanto para la población control como para los pacientes con diagnóstico de LMC, mientras que el SNP CYP3A4*1B (AG en la posición -290) mostró una frecuencia de 0.11 en la población control y 0.25 en los pacientes con LMC.
Efecto de CYP3A4*1B sobre la expansión clonal de células Ph+. Para evaluar si hay diferencias significativas entre el porcentaje de Ph+ y el genotipo CYP3A4*1B, se realizó el análisis estadístico no paramétrico de Kruskal-Wallis. Los resultados muestran que el porcentaje del cromosoma biomarcador Philadelphia no difiere con respecto a cada uno de los diferentes genotipos encontrados para CYP3A4*1B (p=0.7208) (Gráfica 2). Con respecto a la variante alélica CYP3A4*2 se descubrió que no se puede incluir como un factor pronóstico del bio-marcador cromosoma Philadelphia, porque dicho SNP está ausente en la muestra poblacional colombiana analizada (n=194).
Interacción tiempo de tratamiento vs. genotipo vs. efecto en el porcentaje de cromosoma Philadelphia. Para evaluar el efecto de la interacción tiempo-genotipo sobre el porcentaje del biomarcador Philadelphia, se realiza un ANOVA de dos factores, para lo que se prueban previamente, homogeneidad de varianzas con la prueba de Levene's, y normalidad de residuos con la prueba SW-W. De esta manera se evalúa el efecto que tiene cada uno de los factores individualmente, y el efecto de la interacción de los mismos. Los resultados de esta prueba, permiten reconfirmar que la duración del tratamiento presenta un efecto significativo en el porcentaje Ph+, pero por otra parte, muestra que la interacción tiempo-genotipo no tiene un efecto significativo en dicho porcentaje (p= 0.694035) (Gráfica 3).
DISCUSIÓN
El imatinib es una novedosa terapia introducida en Colombia hace aproximadamente 4 años, y las estadísticas de efectividad terapéutica apenas se inician. Pero al igual que en otras partes del mundo, está mostrando que en algunos pacientes, la fase de remisión citogenética inicial, requiere de dosis 50% a 100% mayores a la convencional de 400 mg/día17. En consecuencia es un aspecto farmacológico complejo no sólo por tratarse de una terapia costosa que amerita un uso racional, sino que el aumento empírico de un xenobiótico farmacológico puede resultar tóxico y complicar el pronóstico terapéutico al rebasar umbrales individuales e inciertos de biodisponibilidad.
Simultáneamente, se debe tener en cuenta que en este tipo de neoplasias, buena parte del éxito de la quimioterapia depende de la cinética inicial de erradicación de la población celular maligna. De ahí la necesidad de iniciar con terapias efectivas y en algunos casos agresivas. Pero las bases de esta decisión, hasta la fecha no son claras porque con frecuencia dependen más de la evolución clínica conseguida al inicio de la terapia, y menos de criterios farmacogenéticos, que precisamente se empiezan a dilucidar y que se quizo estudiar.
En esta dirección, el presente estudio, conjuga la información de la farmacogenética y la citogenética al confrontar la genotipificación de marcadores farmaco-relevantes para el metabolismo y/o tratamiento con imatinib, con el monitoreo del clon maligno mediante el biomarcador Philadelphia. En este sentido, los resultados citogenéticos concuerdan con lo esperado para un tratamiento farmacológico efectivo de pacientes con diagnóstico de LMC2. Además, demuestran la importancia del marcador citogenético Ph(+), que sin alcanzar la sensibilidad del monitoreo molecular tipo QRT-PCR, tiene la capacidad de máxima especificidad teórica posible (una célula anormal), en contraste con los marcadores moleculares mencionados basados en alta sensibilidad a partir de poblaciones celular y no de células individuales.
La literatura informa14 el polimorfismo CYP3A4*2 como un SNP que presenta diferencias cuantitativas en cuanto a la actividad enzimática de CYP3A4 y que además puede causar alteraciones en la estructura tridimensional de dicha enzima. Con los resultados obtenidos en este estudio, se puede concluir que CYP3A4*2 no constituye un factor pronóstico adverso para la expansión clonal de células Ph(+) en la población de los pacientes estudiados, porque este SNP no se encontró en esta población, como tampoco en los 164 controles genotipificados. Cabe anotar que los controles de digestión para la enzima de restricción MnlI, dieron positivos en el ADN control (exon 6, gen XPD).
Del presente estudio también se concluye que la presencia del polimorfismo CYP3A4*1B a una frecuencia de 0.11 en la población control y 0.25 en los pacientes, no constituye un riesgo para los individuos con diagnóstico de LMC en la población colombiana analizada, pues a pesar de ser un SNP funcional que afecta la farmacocinética de muchos otros medicamentos antineoplásicos porque se asocia con una disminuida actividad metabólica de la enzima14, no mostró un efecto significativo en la evolución citogenética del biomarcador Philadelphia en los pacientes con LMC. En consecuencia, es razonable concluir que CYP3A4*2 no representa un riesgo para el buen desarrollo de la farmacoterapia con imatinib en personas con leucemia mieloide crónica en nuestra población.
Los valores de frecuencia alélica, 0.11 para CYP3A4*1B y 0.0 para CYP3A4*2 se asimilan a las frecuencias alélicas informadas para estos polimorfismos en caucásicos, como es el caso de 0.096 para CYP3A4*1B13 y 0.027 para CYP3A4*2 en población española14. Lo anterior, sería compatible con el orígen étnico preponderante de la población analizada, en la cual está ampliamente demostrado un componente caucásico que oscila entre 70% y 80%18,19. También es pertinente observar la escasez de estudios de este tipo en poblaciones tri-étnicas como la actual; uno de ellos, realizado en población mexicana, coincide en informar una frecuencia de 0.0 para CYP3A4*213.
Con este acercamiento farmacogenético piloto, se explora y resalta la importancia del análisis de la variabilidad interindividual en la respuesta a la quimioterapia con imatinib. Cada vez es más evidente la sensibilidad diferencial a los medicamentos en personas y poblaciones del mundo, cuyas respuestas por lo general están basadas en dos componentes principales: uno ambiental, como es la co-administración de medicamentos, y otro genético como son el origen étnico y los polimorfismos genómicos que afectan de manera directa la farmacocinética de diversos medicamentos20. Es un concepto biomédico que se está abriendo camino hacia la farmacogenética aplicada, y eventualmente individualizada, al menos para fármacos o xenobióticos metabolizados por enzimas codificadas por genes que contienen variaciones alélicas tipo SNPs de relevancia farmacológica, y presentes en diferentes proporciones en diversas poblaciones.
CONCLUSIONES
Los resultados obtenidos en este estudio muestran un panorama farmacogenético favorable para el tratamiento con imatinib en pacientes colombianos, en relación con el importante metabolizador de medicamentos como es el CYP3A4. Se hace necesario entonces, continuar con la evaluación y búsqueda de otras variaciones alélicas de importancia farmacológica, bien sea en otras enzimas del metabolismo del imatinib o variaciones en el gen de fusión BCR-ABL, todo esto con miras al mejoramiento específico de la terapia farmacológica en LMC, y la búsqueda de co-factores farmacogenéticos individuales.
AGRADECIMIENTOS
Agradecemos a todos los paciente que colaboraron voluntariamente en esta investigación y al Grupo de Genética de Poblaciones y Mutacarcinogénesis, Sede de Investigación Universitaria (SIU). Los autores expresan no tener conflictos de intereses intelectuales ni económicos.
REFERENCIAS
1. Deininger MWN, Goldman JM, Melo JV. The molecular biology of chronic myeloid leukemia. Blood. 2000; 96: 3343-56.
2. Kantarjian H, Sawyers C, Hochhaus A, Guilhot F, Schiffer C, Gambacorti-Passerini C, et al. International STI571 CML Study Group. Hematologic and cytogenetic responses to imatinib mesylate in chronic myelogenous leukemia. N Engl J Med. 2002; 346: 645-52.
3. Graham J, Debiec-Rychter M, Corless CL, Reid R, Davidson R, White JD. imatinib in the management of multiple gastrointestinal stromal tumors associated with a germline KIT K642E mutation. Arch Pathol Lab Med. 2007; 131: 1393-6.
4. Cools J, DeAngelo DJ, Gotlib J, Stover EH, Legare RD, Cortes J, et al. A tyrosine kinase created by fusion of the PDGFRA and FIP1L1 genes as a therapeutic target of imatinib in idiopathic hypereosinophilic syndrome. N Engl J Med. 2003; 348: 1201-14.
5. Carpiuc KT, Stephens JM, Botteman MF, Feng W, Hay JW. A review of the clinical and economic outcomes of imatinib in Philadelphia chromosome-positive acute lymphoblastic leukemia. Expert Opin Pharmacother. 2007; 8: 2775-87.
6. Piccaluga PP, Rondoni M, Paolini S, Rosti G, Martinelli G, Baccarani M. imatinib mesylate in the treatment of hematologic malignancies. Expert Opin Biol Ther. 2007; 7: 1597-611.
7. Druker BJ, Tamura S, Buchdunger E, Ohno S, Segal GM, Fanning S, et al. Effects of a selective inhibitor of the Abl tyrosine kinase on the growth of Bcr-Abl positive cells. Nat Med. 1996; 2: 561-6.
8. Buchdunger E, Zimmermann J, Mett H, Buchdunger E, Meyer T, Muller M, et al. Inhibition of the Abl protein-tyrosine kinase in vitro and in vivo by a 2-phenylaminopyrimidine derivative. Cancer Res. 1996; 56: 100-4.
9. Frye RF, Fitzgerald SM, Lagattuta TF, Hruska MW, Egorin MJ. Effect of St John's wort on imatinib mesylate pharmacokinetics. Clin Pharmacol Ther. 2004; 76: 323-9.
10. Van Schaik RH, De Wildt SN, Van Iperen NM, Uitterlinden AG, Van Den Anker JN, Lindemans J. CYP3A4-V polymorphism detection by PCR-RFLP and its allelic frequency among 199 Dutch Caucasians. Clin Chem. 2000; 46: 1834-5.
11. Plant N, Gibson GG. Evaluation of the toxicological relevance of CYP3A4 induction. Curr Opin Drug Discov Devel. 2003; 6: 50-6.
12. Liu TC, Lin SF, Chen TP, Chang JG. Polymorphism analysis of CYP3A5 in myeloid leukemia. Oncol Report. 2002; 9: 327-9.
13. Reyes-Hernández OD, Arteaga-Illán G, Elizondo G. Detection of CYP3A4*1B and CYP3A4*2 polymorphisms by RFLP. Distribution frequencies in a Mexican population. Clin Genet. 2004; 66: 166-8.
14. Sata F, Sapone A, Elizondo G, Stocker P, Miller VP, Zheng W, et al. CYP3A4 allelic variants amino acid substitutions in exons 7 and 12: Evidence for an allelic variant with altered catalytic activity. Clin Pharmacol Ther. 2000; 67: 48-56.
15. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acid Res. 1988; 16: 1215-7.
16. Camargo M, Cervenka J. Patterns of DNA replication of human chromosomes. II. Replication map and replication model. Am J Hum Genet. 1982; 34: 757-80.
17. Kantarjian HM, Talpaz M, O'Brien S, Giles F, García-Manero G, Faderl S, et al. Dose escalation of imatinib mesylate can overcome resistance to standard-dose therapy in patients with chronic myelogenous leukaemia. Blood. 2003; 101: 473-5.
18. Bravo ML, Valenzuela CY, Arcos-Burgos OM. Polymorphisms and phyletic relationships of the Paisa community from Antioquia (Colombia). Gene Geogr. 1996; 10: 11-7.
19. Carvajal-Carmona LG, Soto ID, Pineda N, Ortíz-Barrientos D, Duque C, Ospina-Duque J, et al. Strong Amerind/white sex bias and a possible Sephardic contribution among the founders of a population in northwest Colombia. Am J Hum Genet. 2000; 67: 1287-95.
20. Potti A, Dressman HK, Bild A, Riedel RF, Chan G, Sayer R, et al. Genomic signatures to guide the use of chemotherapeutics. Nat Med. 2006; 12: 1294-300.